[Déprécié] Tutoriel pour débutant Chainer v1.24.0
avertissement
Cet article est trop vieux pour être ridicule, écrit pour la version finale de Chainer v1 (v1.24.0), qui n'est plus supportée.
** Il y a un article écrit pour la dernière version stable de Chainer v3 en décembre 2017 à ici, il est donc absolument nécessaire de démarrer avec la v1. Sauf si vous avez une situation très particulière, veuillez vous rendre à là-bas maintenant. ** **
Allez !! -> Tutoriel pour débutant Chainer v3
1st Chainer Beginner's Hands-on s'est tenu dans la salle polyvalente du bureau de Preferred Networks à Otemachi. Cet article est un article sur ce que j'ai fait dans cette pratique.
Les matériaux utilisés le jour de la pratique sont résumés dans le référentiel Github suivant.
Le jour de l'événement, 20 serveurs GPU équipés de 4 Pascal TITAN X (80 GPU au total!) Ont été empruntés gratuitement à Sakura Internet, et des travaux pratiques ont été organisés pour que tous les participants puissent les utiliser. Nous aimerions profiter de cette occasion pour remercier Sakura Internet. On dit que Sakura High Thermal Power Computing lancera bientôt un service de location de serveur GPU toutes les heures, donc si vous envisagez d'introduire un environnement GPU, veuillez le vérifier.
Calcul à haute puissance thermique Sakura
Le jour de la mise en route, j'ai commencé par me connecter à chaque nœud de ce Sakura emprunté à haute puissance thermique et installer NVIDIA CUDA, mais dans cet article, j'ai sauté cette partie et résumé de la partie sur l'utilisation de Chainer Je vais continuer.
Veuillez vous référer aux matériaux suivants pour savoir comment construire l'environnement.
Construction de l'environnement sur le serveur de calcul haute température Sakura
Cela peut être utilisé comme procédure de création d'un environnement pour un serveur équipé d'un GPU NVIDIA fonctionnant sous Ubuntu 14.04, à quelques exceptions près.
Passons maintenant au sujet principal. Ce qui suit est un tutoriel écrit en supposant Python 3.4, qui est installé par défaut sur Ubuntu 14.04. Veuillez vous référer aux P.9 et P.11 du document ci-dessus à l'avance pour installer les bibliothèques associées et Chainer lui-même. ** La partie de code suivante et le résultat de sortie qui suit sont supposés être exécutés sur le notebook Jupyter. ** **
Écrivons une boucle d'apprentissage
ici,
- Extraire les données de l'ensemble de données
- Remplissez le modèle
- Utilisez Optimizer pour mettre à jour les paramètres du modèle et exécuter une boucle d'entraînement
Je vais essayer ça. Grâce à ceux-ci, vous pouvez découvrir comment écrire une boucle d'apprentissage sans utiliser Trainer.
1. Préparation de l'ensemble de données
Ici, nous utiliserons une méthode pratique pour utiliser l'ensemble de données MNIST fourni par Chainer. Avec lui, tout est caché, du téléchargement de données à la possibilité de récupérer des données individuelles.
from chainer.datasets import mnist
#Si le jeu de données n'a pas été téléchargé, téléchargez-le également
train, test = mnist.get_mnist(withlabel=True, ndim=1)
#Rend le résultat du dessin graphique à l'aide de matplotlib affiché dans le cahier.
%matplotlib inline
import matplotlib.pyplot as plt
#Exemple de données
x, t = train[0]
plt.imshow(x.reshape(28, 28), cmap='gray')
plt.show()
print('label:', t)
Résultat de sortie:
Downloading from http://yann.lecun.com/exdb/mnist/train-images-idx3-ubyte.gz...
Downloading from http://yann.lecun.com/exdb/mnist/train-labels-idx1-ubyte.gz...
Downloading from http://yann.lecun.com/exdb/mnist/t10k-images-idx3-ubyte.gz...
Downloading from http://yann.lecun.com/exdb/mnist/t10k-labels-idx1-ubyte.gz...
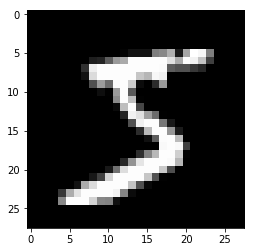
label: 5
2. Créer un itérateur
Créons un «Itérateur» qui récupère un nombre fixe de données de l'ensemble de données et les regroupe pour créer un mini-lot et le renvoyer. Nous allons l'utiliser dans la boucle d'apprentissage qui suit. L'itérateur retournera un nouveau mini-lot avec la méthode next (). En interne, il a des propriétés (ʻis_new_epoch) qui contrôlent combien de tours le jeu de données a été léché (ʻepoch) et si l'itération actuelle est la première itération d'une nouvelle époque.
from chainer import iterators
batchsize = 128
train_iter = iterators.SerialIterator(train, batchsize)
test_iter = iterators.SerialIterator(test, batchsize,
repeat=False, shuffle=False)
À propos d'itérateur
--SerialIterator`, qui est une sorte d'itérateur préparé par Chainer, est l'itérateur le plus simple qui récupère les données dans l'ensemble de données dans l'ordre.
- Prenez l'objet de l'ensemble de données et la taille du lot comme arguments.
--Si vous avez besoin de lire à plusieurs reprises les données de l'objet de jeu de données passé à ce moment pendant de nombreux tours, définissez l'argument
repeatsurTrue, et si vous ne voulez plus récupérer de données après un tour, ceci Soit "Faux". La valeur par défaut est «True». - Si vous passez
Trueà l'argumentshuffle, l'ordre des données extraites de l'ensemble de données sera changé de manière aléatoire pour chaque époque.
Ici, puisque batchsize = 128 est défini, le train_iter qui est l''Iterator pour les données d'apprentissage et le test_iter qui est l''Iterator pour les données de test créées ici sont respectivement 128 données d'image numériques. Sera retourné comme un lot ʻIterator`. [^ Données d'entraînement et données de validation]
3. Définition du modèle
Ici, nous définissons un perceptron simple à trois couches. Il s'agit d'un réseau composé uniquement de couches entièrement connectées. Le nombre d'unités dans la couche intermédiaire est correctement défini sur 100 et la sortie est de 10 classes, définissez-le donc sur 10. En effet, le jeu de données MNIST utilisé ici a 10 étiquettes différentes. Maintenant, jetons un bref coup d'œil aux Link, Function et Chain nécessaires pour définir le modèle.
Lien et fonction
--Chainer distingue chaque couche du réseau neuronal en "Link" et "Function".
- **
Linkest une fonction avec des paramètres. ** ** - **
Functionest une fonction qui n'a pas de paramètres. ** ** - Combinez-les pour décrire le modèle.
- Il y a de nombreuses couches avec des paramètres sous le module
chainer.links. - Il y a de nombreuses couches sans paramètres sous le module
chainer.functions. --Pour les rendre faciles à utiliser
import chainer.links as L
import chainer.functions as F
Il est d'usage de lui donner un alias et de l'utiliser comme L.Convolution2D (...) ou F.relu (...).
Chain
--Chain est une classe ** pour regrouper les couches avec des paramètres = ** Link.
- Avoir des paramètres signifie essentiellement que vous devez les mettre à jour lorsque vous entraînez le modèle (avec des exceptions).
--Ainsi, afin d'obtenir facilement tous les paramètres que ʻOptimizer
devrait mettre à jour pendant l'apprentissage, nous les rassemblerons en un seul endroit avecChain`.
Un modèle défini par l'héritage de Chain
- Le modèle est souvent défini comme une classe qui hérite de la classe
Chain. - Dans ce cas, si vous passez le nom de la couche que vous souhaitez enregistrer sous la forme d'un argument mot-clé et l'objet au constructeur de la classe qui représente le modèle, il sera automatiquement conservé sous la forme trouvée à partir de ʻOptimizer` Je vais le laisser.
- Cela peut également être fait ailleurs en utilisant la méthode ʻadd_link`.
- De plus, il est pratique de définir la méthode
__call__et de décrire le processus de transfert dans celle-ci afin que vous puissiez passer des données au modèle avec l'accesseur()comme un appel de fonction.
Pour exécuter sur GPU
- La classe
Chaina une méthodeto_gpu, et si vous spécifiez un ID GPU dans cet argument, tous les paramètres du modèle seront transférés dans la mémoire de l'ID GPU spécifié. - Ceci est nécessaire pour effectuer des calculs avant / arrière à l'intérieur du modèle sur ce GPU spécifié.
- Si vous ne le faites pas, ces choses seront effectuées sur le processeur.
Définissons maintenant le modèle. Tout d'abord, corrigeons la graine de nombre aléatoire afin que nous puissions reproduire presque le même résultat que cet article. (Si vous voulez garantir plus strictement la reproductibilité du résultat du calcul, vous devez connaître l'option déterministe. Cet article est utile: Pourquoi et la contre-mesure que le résultat change à chaque fois lors de l'utilisation de GPU avec Chainer //qiita.com/TokyoMickey/items/cc8cd43545f2656b1cbd).
import numpy
numpy.random.seed(0)
import chainer
if chainer.cuda.available:
chainer.cuda.cupy.random.seed(0)
Définissons maintenant le modèle, créons un objet et l'envoyons au GPU.
import chainer
import chainer.links as L
import chainer.functions as F
class MLP(chainer.Chain):
def __init__(self, n_mid_units=100, n_out=10):
#Enregistrement des couches avec des paramètres
super(MLP, self).__init__(
l1=L.Linear(None, n_mid_units),
l2=L.Linear(n_mid_units, n_mid_units),
l3=L.Linear(n_mid_units, n_out),
)
def __call__(self, x):
#Écrire le calcul avant la réception des données
h1 = F.relu(self.l1(x))
h2 = F.relu(self.l2(h1))
return self.l3(h2)
gpu_id = 0
model = MLP()
model.to_gpu(gpu_id) #Si vous voulez que le CPU fasse le travail, commentez cette ligne.
NOTE
Ici, la classe «L.Linear» signifie une couche entièrement connectée. Si vous passez None comme premier argument du constructeur, au moment de l'exécution, le moment où les données sont entrées dans cette couche, le nombre d'unités d'entrée requis sera calculé automatiquement, et (n_input) $ \ times $ Créez une matrice de taille n_mid_units et conservez-la comme paramètre. C'est une fonctionnalité utile plus tard, par exemple lorsque vous placez la couche de convolution devant la couche entièrement connectée.
Comme mentionné précédemment, Link a un paramètre, vous pouvez donc accéder à la valeur de ce paramètre. Par exemple, le modèle «MLP» ci-dessus a une couche entièrement connectée nommée «l1» enregistrée. Cette phase entièrement couplée a deux paramètres, "W" et "b". Ceux-ci sont accessibles de l'extérieur. Par exemple, pour accéder à «b», procédez comme suit:
print('La forme du paramètre de polarisation de la première phase entièrement couplée est', model.l1.b.shape)
print('La valeur immédiatement après l'initialisation est', model.l1.b.data)
Résultat de sortie
La forme du paramètre de polarisation de la première phase entièrement couplée est(100,)
La valeur immédiatement après l'initialisation est[ 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0.]
Maintenant, lorsque j'essaye d'accéder à model.l1.W, j'obtiens l'erreur suivante:
AttributeError: 'Linear' object has no attribute 'W'
En effet, dans la définition du modèle ci-dessus, «Aucun» est passé comme premier argument du constructeur du lien «Linéaire», de sorte que la matrice «W» n'est pas allouée avant l'exécution. Il n'existe pas, mais il est connu à l'intérieur de l'objet Linear qu'il existera.
4. Sélection de la méthode d'optimisation
Chainer propose de nombreuses techniques d'optimisation. Ils sont sous le module chainer.optimizers. Ici, nous utilisons ʻoptimizers.SGD, qui est la méthode de descente de gradient la plus simple. Passez le modèle (objet Chain) à l'objet Optimizer en utilisant la méthode setup`. Cela permettra à Optimizer de suivre automatiquement les paramètres du modèle qu'il doit mettre à jour.
Vous pouvez facilement essayer diverses autres méthodes d'optimisation, alors essayez-les et voyez comment les résultats changent. Par exemple, dans chainer.optimizers.SGD ci-dessous, changez la partie SGD en Momentum SGD, RMSprop, Adam, etc. et voyez la différence dans les résultats.
from chainer import optimizers
optimizer = optimizers.SGD(lr=0.01)
optimizer.setup(model)
NOTE
Cette fois, j'ai donné 0,01 $ à l'argument lr dans le constructeur SGD. Cette valeur est connue sous le nom de taux d'apprentissage et est connue comme un ** hyperparamètre ** important qui doit être ajusté pour bien entraîner le modèle pour de bonnes performances.
5. Boucle d'apprentissage
C'est finalement une boucle d'apprentissage. Puisqu'il s'agit d'un problème de classification, nous utiliserons la fonction de perte softmax_cross_entropy pour calculer la valeur de la perte à minimiser.
Dans Chainer, le calcul avant du modèle est effectué à l'aide de Function et Link, le résultat et l'étiquette de réponse correcte sont passés à la fonction de perte qui est une sorte de Function et renvoie la valeur scalaire, et la perte est calculée. Comme tout autre Link ou Function, il renvoie un objet Variable. Étant donné que l'objet Variable contient une référence pour revenir au processus de calcul précédent dans le sens inverse, appelez simplement la méthode Variable.backward () et le calcul à partir de là sera effectué automatiquement. Il remonte dans le processus et calcule le gradient des paramètres utilisés dans le calcul effectué au milieu.
En d'autres termes, les quatre éléments suivants sont exécutés dans une boucle d'apprentissage.
- Passez les données au modèle et obtenez la sortie
y - À l'aide de «y» et de la bonne étiquette «t», calculez la valeur de la perte à minimiser avec la fonction «softmax_cross_entropy».
- Appelez la méthode «backward» de la «variable de sortie» de la fonction «softmax_cross_entropy» pour donner aux paramètres à l'intérieur du modèle la propriété «grad» (qui est le gradient utilisé pour mettre à jour les paramètres).
- Appelez la méthode ʻupdate
d'Optimizer et mettez à jour tous les paramètres en utilisant legrad` calculé en 3.
c'est tout. Si vous travaillez sur un problème de régression simple au lieu d'un problème de classification, vous pouvez utiliser F.mean_squared_error au lieu de F.softmax_cross_entropy. En outre, Chainer fournit diverses fonctions de perte pour traiter divers paramètres de problème. Vous pouvez voir la liste ici: Fonctions de perte.
Maintenant, écrivons une boucle d'entraînement.
import numpy as np
from chainer.dataset import concat_examples
from chainer.cuda import to_cpu
max_epoch = 10
while train_iter.epoch < max_epoch:
# ----------1 Itération d'apprentissage----------
train_batch = train_iter.next()
x, t = concat_examples(train_batch, gpu_id)
#Calcul de la valeur prédite
y = model(x)
#Calcul des pertes
loss = F.softmax_cross_entropy(y, t)
#Calcul du gradient
model.cleargrads()
loss.backward()
#Mise à jour des paramètres
optimizer.update()
# ---------------Jusque là----------------
#Mesurer la précision de la prédiction des données de validation à la fin de chaque époque,
#Vérifions que les performances de généralisation du modèle sont améliorées
if train_iter.is_new_epoch: #Quand 1 époque est terminée
#Affichage de la perte
print('epoch:{:02d} train_loss:{:.04f} '.format(
train_iter.epoch, float(to_cpu(loss.data))), end='')
test_losses = []
test_accuracies = []
while True:
test_batch = test_iter.next()
x_test, t_test = concat_examples(test_batch, gpu_id)
#Transfert des données de test
y_test = model(x_test)
#Calculer la perte
loss_test = F.softmax_cross_entropy(y_test, t_test)
test_losses.append(to_cpu(loss_test.data))
#Calculer la précision
accuracy = F.accuracy(y_test, t_test)
accuracy.to_cpu()
test_accuracies.append(accuracy.data)
if test_iter.is_new_epoch:
test_iter.epoch = 0
test_iter.current_position = 0
test_iter.is_new_epoch = False
test_iter._pushed_position = None
break
print('val_loss:{:.04f} val_accuracy:{:.04f}'.format(
np.mean(test_losses), np.mean(test_accuracies)))
Résultat de sortie
epoch:01 train_loss:0.7828 val_loss:0.8276 val_accuracy:0.8167
epoch:02 train_loss:0.3672 val_loss:0.4564 val_accuracy:0.8826
epoch:03 train_loss:0.3069 val_loss:0.3702 val_accuracy:0.8976
epoch:04 train_loss:0.3333 val_loss:0.3307 val_accuracy:0.9078
epoch:05 train_loss:0.3308 val_loss:0.3079 val_accuracy:0.9129
epoch:06 train_loss:0.3210 val_loss:0.2909 val_accuracy:0.9162
epoch:07 train_loss:0.2977 val_loss:0.2781 val_accuracy:0.9213
epoch:08 train_loss:0.2760 val_loss:0.2693 val_accuracy:0.9232
epoch:09 train_loss:0.1762 val_loss:0.2566 val_accuracy:0.9263
epoch:10 train_loss:0.2444 val_loss:0.2479 val_accuracy:0.9284
Si vous regardez le val_accuracy, vous vous retrouvez avec 0,9286 $ pour 10 époques. Les nombres manuscrits peuvent désormais être classés avec une précision d'environ 93%.
6. Enregistrez le modèle entraîné
Chainer a deux fonctionnalités de sérialisation. L'une consiste à enregistrer le modèle au format HDF5 et l'autre à enregistrer le modèle au format NPZ de NumPy. Cette fois, au lieu de HDF5, qui nécessite l'installation de bibliothèques supplémentaires, nous enregistrerons le modèle au format NPZ en utilisant la fonction de sérialisation fournie par la fonction standard NumPy.
from chainer import serializers
serializers.save_npz('my_mnist.model', model)
#Assurez-vous qu'il est correctement enregistré
%ls -la my_mnist.model
\ * La dernière ligne ne fonctionne que sur le notebook Jupyter.
Résultat de sortie
-rw-rw-r-- 1 ubuntu ubuntu 333853 Mar 29 16:51 my_mnist.model
7. Charger et déduire le modèle enregistré
Chargeons le fichier NPZ que nous venons d'enregistrer et laissons le réseau prédire l'étiquette des données de test. Étant donné que les paramètres sont enregistrés dans le fichier NPZ, créez d'abord un objet du modèle avec la logique du calcul avant et écrasez les paramètres avec la valeur de la NPZ enregistrée précédemment pour restaurer l'état du modèle immédiatement après l'entraînement. Je vais.
#Créez d'abord un objet du même modèle
infer_model = MLP()
#Charger les paramètres enregistrés dans cet objet
serializers.load_npz('my_mnist.model', infer_model)
#Envoyez le modèle au GPU pour le calcul sur le GPU
infer_model.to_gpu(gpu_id)
#données de test
x, t = test[0]
plt.imshow(x.reshape(28, 28), cmap='gray')
plt.show()
print('label:', t)
Résultat de sortie

label: 7
J'ai essayé d'afficher les données de test que le modèle inférera désormais. Voici un exemple de réalisation d'une inférence pour cette image.
from chainer.cuda import to_gpu
#Réalisez-le sous la forme d'un mini-lot (ici, il s'agit d'un mini-lot de taille 1,
#Il est également possible de déduire collectivement plusieurs mini-lots de taille n)
print(x.shape, end=' -> ')
x = x[None, ...]
print(x.shape)
#Les données sont également envoyées sur le GPU pour le calcul sur le GPU
x = to_gpu(x, 0) #Si vous voulez le faire sur le CPU, commentez ici.
#Passer à la fonction avant du modèle
y = infer_model(x)
#Comme il sort au format variable, retirez le contenu
y = y.data
#Envoyer le résultat à la CPU
y = to_cpu(y) #Si vous voulez le faire sur le CPU, commentez ici.
#Afficher l'index maximum
pred_label = y.argmax(axis=1)
print('predicted label:', pred_label[0])
Résultat de sortie
(784,) -> (1, 784)
predicted label: 7
Utilisons Trainer
Trainer élimine le besoin d'écrire explicitement des boucles d'apprentissage. De plus, diverses extensions pratiques facilitent la visualisation et l'enregistrement des journaux.
1. Préparation de l'ensemble de données
from chainer.datasets import mnist
train, test = mnist.get_mnist()
2. Préparation de l'itérateur
from chainer import iterators
batchsize = 128
train_iter = iterators.SerialIterator(train, batchsize)
test_iter = iterators.SerialIterator(test, batchsize, False, False)
3. Préparation du modèle
Ici, nous utiliserons le même modèle qu'avant.
import chainer
import chainer.links as L
import chainer.functions as F
class MLP(chainer.Chain):
def __init__(self, n_mid_units=100, n_out=10):
super(MLP, self).__init__(
l1=L.Linear(None, n_mid_units),
l2=L.Linear(n_mid_units, n_mid_units),
l3=L.Linear(n_mid_units, n_out),
)
def __call__(self, x):
h1 = F.relu(self.l1(x))
h2 = F.relu(self.l2(h1))
return self.l3(h2)
gpu_id = 0
model = MLP()
model.to_gpu(gpu_id) #Si vous utilisez un processeur, commentez ici.
4. Préparation du programme de mise à jour
Trainer est une classe qui rassemble tout ce dont vous avez besoin pour apprendre ensemble. Trainer et ses classes d'utilité internes, modèles, classes de jeux de données, etc. ont la relation suivante.

Lors de la création d'un objet Trainer, vous ne transmettez que ʻUpdater, mais ʻUpdater a ʻIterator et ʻOptimizer à l'intérieur. Vous pouvez accéder à l'ensemble de données depuis ʻIterator, et ʻOptimizer contient une référence au modèle à l'intérieur, de sorte que vous pouvez mettre à jour les paramètres du modèle. Autrement dit, ʻUpdater` est en interne
- Extraire les données de l'ensemble de données (Iterator)
- Passez-le au modèle et calculez la perte (Model = Optimizer.target)
- Mettre à jour les paramètres du modèle à l'aide de l'Optimizer (Optimizer)
Cela signifie que vous pouvez faire la partie principale de la série d'apprentissage. Créons maintenant un objet ʻUpdater`.
from chainer import optimizers
from chainer import training
max_epoch = 10
gpu_id = 0
#Enveloppez le modèle dans le classificateur et incluez le calcul des pertes, etc. dans le modèle
model = L.Classifier(model)
model.to_gpu(gpu_id) #Si vous utilisez le processeur, commentez cette ligne.
#Sélection de la méthode d'optimisation
optimizer = optimizers.SGD()
optimizer.setup(model)
#Passer Iterator et Optimizer à Updater
updater = training.StandardUpdater(train_iter, optimizer, device=gpu_id)
NOTE
Maintenant, nous passons l'objet modèle défini ci-dessus à L.Classifier pour en faire une nouvelleChain. «L.Classifier» est une classe qui hérite de «Chain» et enregistre la «Chain» passée dans une propriété appelée «prédicteur». Lorsque vous passez des données et une étiquette avec l'accesseur (), __call__ est exécuté à l'intérieur, d'abord les données passées sont passées à travers prédicteur, sa sortie y et __call__ avec les données. Passe l'étiquette passée à la fonction de perte spécifiée par l'argument lossfun du constructeur et renvoie sa sortie Variable. lossfun est spécifié dans softmax_cross_entropy par défaut.
StandardUpdater est la classe la plus simple pour effectuer le traitement dont Updater est en charge comme décrit ci-dessus. En plus de cela, «Parallel Updater», etc. pour l'utilisation de plusieurs GPU sont préparés.
5. Paramètres du formateur
Enfin, définissez le Trainer. Lors de la création d'un objet Trainer, seul l'objet ʻUpdater créé précédemment est requis, mais le deuxième argument stop_trigger indique quand terminer la formation (longueur ,, Si vous donnez un taple sous la forme d'unité) , vous pouvez automatiquement terminer l'apprentissage au moment spécifié. La longueur peut être n'importe quel entier et l'unité peut être `` 'epoch' 'ou `` iteration' '. Si vous ne spécifiez pas stop_trigger`, l'apprentissage ne s'arrêtera pas automatiquement.
#Passer le programme de mise à jour au formateur
trainer = training.Trainer(updater, (max_epoch, 'epoch'),
out='mnist_result')
Dans l'argument ʻout, le répertoire pour enregistrer le fichier journal et le fichier image du graphe décrivant le processus de changement de perte est spécifié à l'aide de l''Extension décrite ci-dessous.
6. Ajouter une extension au formateur
L'avantage d'utiliser «Trainer» est
- Enregistrer automatiquement les journaux dans un fichier (
LogReport) --Afficher régulièrement des informations telles que la perte sur le terminal (PrintReport) - Visualisez périodiquement la perte sous forme de graphique et enregistrez-la sous forme d'image (
PlotReport) - Sérialiser automatiquement le modèle et l'état de l'optimiseur sur une base régulière (
snapshot/snapshot_object) --Affiche une barre de progression indiquant la progression de l'apprentissage (ProgressBar) --Enregistrer la structure du modèle au format de points Graphviz (dump_graph)
Il y a un point que vous pouvez facilement utiliser diverses fonctions pratiques telles que. Pour profiter de ces fonctionnalités, transmettez simplement l'objet ʻExtension que vous voulez ajouter à l'objet Trainer en utilisant la méthode ʻextend. Maintenant, ajoutons en fait quelques «extensions».
from chainer.training import extensions
trainer.extend(extensions.LogReport())
trainer.extend(extensions.snapshot(filename='snapshot_epoch-{.updater.epoch}'))
trainer.extend(extensions.snapshot_object(model.predictor, filename='model_epoch-{.updater.epoch}'))
trainer.extend(extensions.Evaluator(test_iter, model, device=gpu_id))
trainer.extend(extensions.PrintReport(['epoch', 'main/loss', 'main/accuracy', 'validation/main/loss', 'validation/main/accuracy', 'elapsed_time']))
trainer.extend(extensions.PlotReport(['main/loss', 'validation/main/loss'], x_key='epoch', file_name='loss.png'))
trainer.extend(extensions.PlotReport(['main/accuracy', 'validation/main/accuracy'], x_key='epoch', file_name='accuracy.png'))
trainer.extend(extensions.dump_graph('main/loss'))
LogReport
Il totalise automatiquement la «perte», la «précision», etc. pour chaque «epoch» et «itération», et les enregistre dans le répertoire de sortie spécifié par l'argument «out» de «Trainer» avec le nom de fichier «log».
snapshot
L'objet Trainer est enregistré dans le répertoire de sortie spécifié par l'argument ʻout de Trainer au moment spécifié (par défaut, toutes les 1 époques). Comme mentionné ci-dessus, l'objet Trainer a ʻUpdater, et ʻOptimizer et le modèle y est conservé, donc si vous prenez un instantané avec cette ʻExtension`, vous pouvez revenir à l'apprentissage ou apprendre. L'inférence utilisant un modèle terminé est possible même après la fin de l'apprentissage.
snapshot_object
Cependant, si vous enregistrez l'intégralité du Trainer, il peut souvent être fastidieux de ne récupérer que le modèle à l'intérieur. Par conséquent, seuls les objets spécifiés à l'aide de snapshot_object (ici, le modèle enveloppé dans Classifier) doivent être enregistrés séparément de Trainer. Classifier est uneChain qui contient l'objet Chain passé au premier argument comme sa propre propriété appelée prédicteur et calcule la perte, et Classifier n'a pas de paramètres autres que le modèle en premier lieu. Par conséquent, ici, «model.predictor» est spécifié comme cible de stockage en prévision de l'utilisation du modèle entraîné pour l'inférence plus tard.
dump_graph
Enregistre le graphique de calcul qui peut être tracé à partir de l'objet Variable spécifié au format de points Graphviz. La destination de sauvegarde est le répertoire de sortie spécifié par l'argument ʻout de Trainer`.
Evaluator
En passant «Itérateur» de l'ensemble de données d'évaluation et de l'objet du modèle utilisé pour l'apprentissage, le modèle en cours d'apprentissage est évalué en utilisant l'ensemble de données d'évaluation au moment spécifié.
PrintReport
Renvoie les valeurs agrégées par Reporter à la sortie standard. A ce moment, la valeur à afficher est donnée sous forme de liste.
PlotReport
Dessine la transition de la valeur spécifiée dans la liste d'arguments sur le graphique à l'aide de la bibliothèque matplotlib, et l'enregistre en tant qu'image dans le répertoire de sortie avec le nom de fichier spécifié par l'argument nom_fichier.
En plus de celles introduites ici, ces «extensions» ont plusieurs options, telles que la possibilité de spécifier quand opérer individuellement avec le «déclencheur», permettant plus de flexibilité dans la combinaison. Voir la documentation officielle pour plus d'informations: Extensions du formateur.
7. Commencez à apprendre
Pour commencer l'apprentissage, il suffit d'appeler la méthode de l'objet Trainer`` run.
trainer.run()
Résultat de sortie
epoch main/loss main/accuracy validation/main/loss validation/main/accuracy elapsed_time
1 1.6035 0.61194 0.797731 0.833564 2.98546
2 0.595589 0.856793 0.452023 0.88123 5.74528
3 0.4241 0.885944 0.368583 0.897943 8.34872
4 0.367762 0.897152 0.33103 0.905756 11.4449
5 0.336136 0.904967 0.309321 0.912282 14.2671
6 0.314134 0.910464 0.291451 0.914557 17.0762
7 0.297581 0.914879 0.276472 0.920985 19.8298
8 0.283512 0.918753 0.265166 0.923655 23.2033
9 0.271917 0.922125 0.254976 0.926523 26.1452
10 0.260754 0.925123 0.247672 0.927413 29.3136
J'ai pu obtenir le même résultat avec des informations de journal riches et des graphiques comme celui montré ci-dessous, avec un code plus court que si j'avais écrit la boucle d'apprentissage sur laquelle j'ai travaillé au début.
Vérifions immédiatement le graphique des pertes enregistrées.
from IPython.display import Image
Image(filename='mnist_result/loss.png')
\ * Cette partie doit être exécutée sur le notebook Jupyter pour obtenir les résultats suivants.
Résultat de sortie

Regardons également le graphique de précision.
Image(filename='mnist_result/accuracy.png')
Résultat de sortie
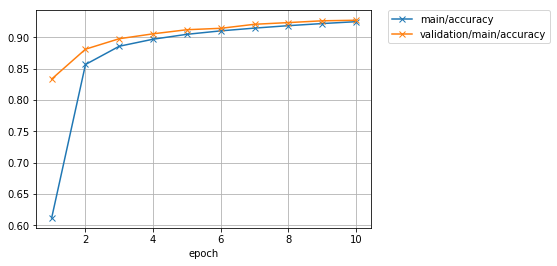
Si vous continuez à apprendre un peu plus, il y a toujours une atmosphère où vous pouvez améliorer un peu la précision.
Ensuite, utilisons Graphviz pour visualiser la sortie du graphique de calcul par ʻExtension appelée dump_graph`.
%%bash
dot -Tpng mnist_result/cg.dot -o mnist_result/cg.png
\ * Ici, nous utilisons Cell magic qui utilise la commande bash sur le notebook Jupyter. La commande sur la deuxième ligne elle-même est une commande shell normale.
Image(filename='mnist_result/cg.png')
Résultat de sortie

De haut en bas, vous pouvez voir à quelle «fonction» les données et les paramètres ont été passés pour le calcul et la «variable» pour la perte a été sortie.
8. Inférer avec un modèle entraîné
import numpy as np
from chainer import serializers
from chainer.cuda import to_gpu
from chainer.cuda import to_cpu
model = MLP()
serializers.load_npz('mnist_result/model_epoch-10', model)
model.to_gpu(gpu_id)
%matplotlib inline
import matplotlib.pyplot as plt
x, t = test[0]
plt.imshow(x.reshape(28, 28), cmap='gray')
plt.show()
print('label:', t)
x = to_gpu(x[None, ...])
y = model(x)
y = to_cpu(y.data)
print('predicted_label:', y.argmax(axis=1)[0])
Résultat de sortie

label: 7
predicted_label: 7
J'ai pu répondre correctement.
Écrivons un nouveau réseau
Ici, au lieu d'utiliser le jeu de données MNIST, essayez d'écrire vous-même divers modèles et de vivre le flux d'essais et d'erreurs en utilisant une petite image couleur de taille 32x32 appelée CIFAR10 étiquetée avec l'une des 10 classes. Je vais.
| airplane | automobile | bird | cat | deer | dog | frog | horse | ship | truck |
|---|---|---|---|---|---|---|---|---|---|
 |
 |
 |
 |
 |
 |
 |
 |
 |
 |
1. Définition du modèle
Le modèle est défini en héritant de la classe Chain. Ici, définissons un réseau avec une couche de convolution au lieu du réseau constitué uniquement des couches entièrement connectées que nous avons essayées précédemment. Ce modèle comporte trois couches convolutives, suivies de deux couches entièrement connectées.
Le modèle est principalement défini en définissant deux méthodes.
- Définissez les couches qui composent le modèle avec le constructeur
__init__
- À ce stade, une couche avec des paramètres de cible d'optimisation qui peuvent être capturés à partir de ʻOptimizer
en passant un objetLinkqui compose le modèle comme argument de mot-clé en utilisantsuperau constructeur de la classe parent (Chain`). Peut être ajouté au modèle.
- Écrivez le calcul Forward dans la méthode
__call__appelée par l'accesseur` () ʻ qui reçoit les données.
import chainer
import chainer.functions as F
import chainer.links as L
class MyModel(chainer.Chain):
def __init__(self, n_out):
super(MyModel, self).__init__(
conv1=L.Convolution2D(None, 32, 3, 3, 1),
conv2=L.Convolution2D(32, 64, 3, 3, 1),
conv3=L.Convolution2D(64, 128, 3, 3, 1),
fc4=L.Linear(None, 1000),
fc5=L.Linear(1000, n_out)
)
def __call__(self, x):
h = F.relu(self.conv1(x))
h = F.relu(self.conv2(h))
h = F.relu(self.conv3(h))
h = F.relu(self.fc4(h))
h = self.fc5(h)
return h
2. Apprentissage
Ici, définissez la fonction train afin de pouvoir facilement entraîner un autre modèle avec les mêmes paramètres ultérieurement. c'est,
- Objet modèle --Taille du lot
- ID GPU à utiliser --Nombre d'époques pour terminer l'apprentissage --Dataset objet
Si vous réussissez, c'est une fonction qui entraîne le modèle en interne à l'aide de l'ensemble de données passé à l'aide de Trainer et renvoie le modèle dans l'état où la formation est terminée.
Utilisons cette fonction train pour entraîner le modèle MyModel défini ci-dessus.
from chainer.datasets import cifar
from chainer import iterators
from chainer import optimizers
from chainer import training
from chainer.training import extensions
def train(model_object, batchsize=64, gpu_id=0, max_epoch=20, train_dataset=None, test_dataset=None):
# 1. Dataset
if train_dataset is None and test_dataset is None:
train, test = cifar.get_cifar10()
else:
train, test = train_dataset, test_dataset
# 2. Iterator
train_iter = iterators.SerialIterator(train, batchsize)
test_iter = iterators.SerialIterator(test, batchsize, False, False)
# 3. Model
model = L.Classifier(model_object)
if gpu_id >= 0:
model.to_gpu(gpu_id)
# 4. Optimizer
optimizer = optimizers.Adam()
optimizer.setup(model)
# 5. Updater
updater = training.StandardUpdater(train_iter, optimizer, device=gpu_id)
# 6. Trainer
trainer = training.Trainer(updater, (max_epoch, 'epoch'), out='{}_cifar10_result'.format(model_object.__class__.__name__))
# 7. Evaluator
class TestModeEvaluator(extensions.Evaluator):
def evaluate(self):
model = self.get_target('main')
model.train = False
ret = super(TestModeEvaluator, self).evaluate()
model.train = True
return ret
trainer.extend(extensions.LogReport())
trainer.extend(TestModeEvaluator(test_iter, model, device=gpu_id))
trainer.extend(extensions.PrintReport(['epoch', 'main/loss', 'main/accuracy', 'validation/main/loss', 'validation/main/accuracy', 'elapsed_time']))
trainer.extend(extensions.PlotReport(['main/loss', 'validation/main/loss'], x_key='epoch', file_name='loss.png'))
trainer.extend(extensions.PlotReport(['main/accuracy', 'validation/main/accuracy'], x_key='epoch', file_name='accuracy.png'))
trainer.run()
del trainer
return model
model = train(MyModel(10), gpu_id=0) #Lors de l'exécution sur le CPU`gpu_id=-1`Veuillez préciser.
Résultat de sortie
epoch main/loss main/accuracy validation/main/loss validation/main/accuracy elapsed_time
1 1.53309 0.444293 1.29774 0.52707 5.2449
2 1.21681 0.56264 1.18395 0.573746 10.6833
3 1.06828 0.617358 1.10173 0.609773 16.0644
4 0.941792 0.662132 1.0695 0.622611 21.2535
5 0.832165 0.703345 1.0665 0.624104 26.4523
6 0.729036 0.740257 1.0577 0.64371 31.6299
7 0.630143 0.774208 1.07577 0.63953 36.798
8 0.520787 0.815541 1.15054 0.639431 42.1951
9 0.429535 0.849085 1.23832 0.6459 47.3631
10 0.334665 0.882842 1.3528 0.633061 52.5524
11 0.266092 0.90549 1.44239 0.635251 57.7396
12 0.198057 0.932638 1.6249 0.6249 62.9918
13 0.161151 0.944613 1.76964 0.637241 68.2177
14 0.138705 0.952145 1.98031 0.619725 73.4226
15 0.122419 0.957807 2.03002 0.623806 78.6411
16 0.109989 0.962148 2.08948 0.62281 84.3362
17 0.105851 0.963675 2.31344 0.617237 89.5656
18 0.0984753 0.966289 2.39499 0.624801 95.1304
19 0.0836834 0.970971 2.38215 0.626791 100.36
20 0.0913404 0.96925 2.46774 0.61873 105.684
L'apprentissage dure jusqu'à 20 époques. Jetons un coup d'œil au graphique de perte et de précision.
Image(filename='MyModel_cifar10_result/loss.png')

Image(filename='MyModel_cifar10_result/accuracy.png')

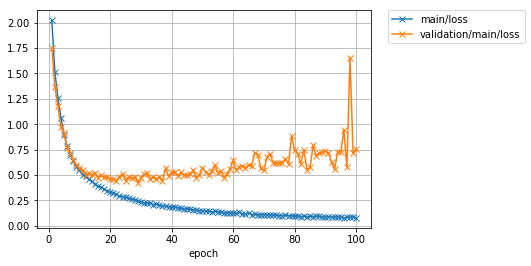
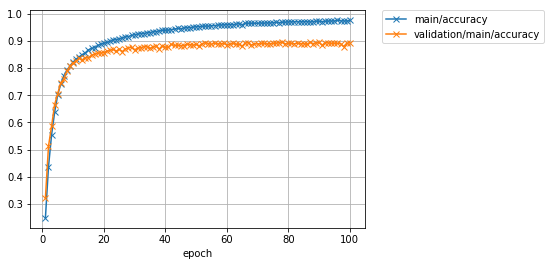
La précision («principale / précision») des données d'entraînement a atteint environ 97%, mais la perte («validation / principale / perte») des données de test a plutôt augmenté à chaque progression de l'itération. En outre, la précision des données de test («validation / principal / précision») a culminé à environ 62%. Il semble que le ** modèle soit surajusté aux données d'entraînement ** car les données d'entraînement ont une bonne précision, mais les données de test ne le sont pas.
3. Prédiction à l'aide d'un modèle entraîné
La précision du test était d'environ 62%, mais essayons d'utiliser ce modèle entraîné pour classer certaines images de test.
%matplotlib inline
import matplotlib.pyplot as plt
cls_names = ['airplane', 'automobile', 'bird', 'cat', 'deer',
'dog', 'frog', 'horse', 'ship', 'truck']
def predict(model, image_id):
_, test = cifar.get_cifar10()
x, t = test[image_id]
model.to_cpu()
y = model.predictor(x[None, ...]).data.argmax(axis=1)[0]
print('predicted_label:', cls_names[y])
print('answer:', cls_names[t])
plt.imshow(x.transpose(1, 2, 0))
plt.show()
for i in range(10, 15):
predict(model, i)
Résultat de sortie
predicted_label: dog
answer: airplane

predicted_label: truck
answer: truck

predicted_label: bird
answer: dog

predicted_label: horse
answer: horse

predicted_label: truck
answer: truck

Certains étaient bien catégorisés, d'autres non. Même si la réponse correcte peut être obtenue dans près de 100 prises de vue sur l'ensemble de données utilisé pour l'apprentissage du modèle, elle n'a aucun sens à moins que les données inconnues, c'est-à-dire l'image dans l'ensemble de données de test, puissent être prédites avec une grande précision [^ NN]. On dit que la précision des données de test est liée à la ** performance de généralisation ** du modèle.
Comment concevoir et former un modèle avec des performances de généralisation élevées?
4. Définissons un modèle plus profond
Définissons maintenant un modèle avec plus de couches que le modèle ci-dessus. Ici, nous définirons un réseau alvéolaire à une couche comme «ConvBlock» et un réseau entièrement connecté à une couche comme «LinearBlock», et définirons un grand réseau en empilant plusieurs d'entre eux séquentiellement.
Définir les composants
Tout d'abord, définissons ConvBlock et LinearBlock, qui sont les composants du grand réseau que nous visons.
class ConvBlock(chainer.Chain):
def __init__(self, n_ch, pool_drop=False):
w = chainer.initializers.HeNormal()
super(ConvBlock, self).__init__(
conv=L.Convolution2D(None, n_ch, 3, 1, 1,
nobias=True, initialW=w),
bn=L.BatchNormalization(n_ch)
)
self.train = True
self.pool_drop = pool_drop
def __call__(self, x):
h = F.relu(self.bn(self.conv(x)))
if self.pool_drop:
h = F.max_pooling_2d(h, 2, 2)
h = F.dropout(h, ratio=0.25, train=self.train)
return h
class LinearBlock(chainer.Chain):
def __init__(self):
w = chainer.initializers.HeNormal()
super(LinearBlock, self).__init__(
fc=L.Linear(None, 1024, initialW=w))
self.train = True
def __call__(self, x):
return F.dropout(F.relu(self.fc(x)), ratio=0.5, train=self.train)
ConvBlock est défini comme un modèle qui hérite de Chain. Il a une couche de convolution et une couche de normalisation par lots avec des paramètres, nous les enregistrons donc dans le constructeur. Dans la méthode __call__, tout en leur passant des données, la fonction d'activation ReLU est appliquée, et lorsque pool_drop est passé au constructeur avec True, les fonctions Max Pooling et Dropout sont appliquées. C'est un petit réseau.
Dans Chainer, le code de calcul avant lui-même écrit en Python représente la structure du modèle. En d'autres termes, la couche traversée par les données lors de l'exécution définit le réseau lui-même. Cela facilite l'écriture de réseaux comprenant des branches comme décrit ci-dessus et permet des définitions de réseau flexibles, simples et hautement lisibles. Il s'agit d'une fonctionnalité appelée ** Define-by-Run **.
Définition d'un grand réseau
Ensuite, empilons ces petits réseaux en tant que composants pour définir un grand réseau.
class DeepCNN(chainer.ChainList):
def __init__(self, n_output):
super(DeepCNN, self).__init__(
ConvBlock(64),
ConvBlock(64, True),
ConvBlock(128),
ConvBlock(128, True),
ConvBlock(256),
ConvBlock(256),
ConvBlock(256),
ConvBlock(256, True),
LinearBlock(),
LinearBlock(),
L.Linear(None, n_output)
)
self._train = True
@property
def train(self):
return self._train
@train.setter
def train(self, val):
self._train = val
for c in self.children():
c.train = val
def __call__(self, x):
for f in self.children():
x = f(x)
return x
La classe que nous utilisons ici est «ChainList». Cette classe est une classe qui hérite de «Chain» et est utile lors de la définition d'un réseau qui appelle plusieurs «Link» et «Chain» en séquence. Les modèles définis en héritant de ChainList peuvent passer des objets ** Link ou Chain comme arguments normaux au lieu de ** arguments de mot-clé lors de l'appel du constructeur de la classe parent. Et ceux-ci peuvent être récupérés ** dans l'ordre où ils ont été enregistrés ** par la méthode ** self.children () **.
Cette fonctionnalité facilite l'écriture de calculs en aval. À partir de la liste des composants renvoyés par ** self.children () **, les composants sont extraits dans l'ordre par l'instruction for, et le calcul de réseau partiel extrait est appliqué à l'entrée d'origine «x». Si vous remplacez «x» par cette sortie dans l'ordre, vous pouvez appliquer une série de «Link» ou «Chain» dans le même ordre que celui enregistré dans la classe parente dans le constructeur. Par conséquent, il est utile pour définir un grand réseau représenté par l'application de réseaux partiels séquentiels.
Passons maintenant à l'apprentissage. Puisqu'il y a beaucoup de paramètres cette fois, réglez le nombre d'époques pour arrêter l'apprentissage à 100.
model = train(DeepCNN(10), max_epoch=100)
Résultat de sortie
epoch main/loss main/accuracy validation/main/loss validation/main/accuracy elapsed_time
1 2.05147 0.242887 1.71868 0.340764 14.8099
2 1.5242 0.423816 1.398 0.48537 29.12
3 1.24906 0.549096 1.12884 0.6042 43.4423
4 0.998223 0.652649 0.937086 0.688495 58.291
5 0.833486 0.720009 0.796678 0.73756 73.4144
.
.
.
95 0.0454193 0.987616 0.815549 0.863555 1411.86
96 0.0376641 0.990057 0.878458 0.873109 1426.85
97 0.0403836 0.98953 0.849209 0.86465 1441.19
98 0.0369386 0.989677 0.919462 0.873905 1456.04
99 0.0361681 0.990677 0.88796 0.86873 1470.46
100 0.0383634 0.988676 0.92344 0.869128 1484.91
(Le journal étant long, le milieu est omis.)
L'apprentissage est terminé. Jetons un coup d'œil aux graphiques de perte et de précision.
Image(filename='DeepCNN_cifar10_result/loss.png')

Image(filename='DeepCNN_cifar10_result/accuracy.png')

Vous pouvez voir que la précision des données de test s'est considérablement améliorée par rapport à la fois précédente. La précision, qui était d'environ 62%, est passée à environ 87%. Cependant, les derniers résultats de recherche ont atteint près de 97%. Afin d'améliorer encore la précision, non seulement l'amélioration du modèle comme cela a été fait cette fois, mais aussi l'opération d'augmentation artificielle des données d'apprentissage (Augmentation des données) et l'opération d'intégration des sorties de plusieurs modèles en une seule sortie (Ensemble). Et ainsi de suite, diverses idées peuvent être envisagées.
Écrivons une classe d'ensemble de données
Ici, j'écrirai moi-même la classe du jeu de données en utilisant la fonction pour acquérir les données de CIFAR10 déjà préparées dans Chainer. Chainer requiert que la classe qui représente l'ensemble de données ait les fonctionnalités suivantes:
--__len__ méthode qui renvoie le nombre de données dans l'ensemble de données
- La méthode
get_examplequi renvoie les données ou la paire données / étiquette qui correspond à ʻi` passée en argument.
La fonctionnalité requise pour d'autres ensembles de données peut être fournie en héritant de la classe chainer.dataset.DatasetMixin. Ici, créons une classe d'ensemble de données avec la fonction d'augmentation de données en héritant de la classe DatasetMixin.
1. Ecrire une classe de jeu de données CIFAR10
import numpy as np
from chainer import dataset
from chainer.datasets import cifar
class CIFAR10Augmented(dataset.DatasetMixin):
def __init__(self, train=True):
train_data, test_data = cifar.get_cifar10()
if train:
self.data = train_data
else:
self.data = test_data
self.train = train
self.random_crop = 4
def __len__(self):
return len(self.data)
def get_example(self, i):
x, t = self.data[i]
if self.train:
x = x.transpose(1, 2, 0)
h, w, _ = x.shape
x_offset = np.random.randint(self.random_crop)
y_offset = np.random.randint(self.random_crop)
x = x[y_offset:y_offset + h - self.random_crop,
x_offset:x_offset + w - self.random_crop]
if np.random.rand() > 0.5:
x = np.fliplr(x)
x = x.transpose(2, 0, 1)
return x, t
Cette classe est pour chacune des données CIFAR10
--Cadrer la zone 28x28 au hasard à partir de la taille 32x32 --Inverser la gauche et la droite avec 1/2 chance
Nous faisons le traitement. On sait que l'augmentation de la variation des données d'apprentissage d'une manière pseudo en ajoutant de telles opérations est utile pour supprimer le surajustement. En plus de ces opérations, divers traitements tels que la conversion qui change la couleur de l'image, la rotation aléatoire et la conversion affine ont été proposés pour augmenter le nombre de données d'apprentissage d'une manière pseudo.
Si vous voulez écrire vous-même la partie d'acquisition de données, passez le chemin du dossier image et le chemin du fichier texte avec l'étiquette correspondant au nom du fichier au constructeur et conservez-le comme propriété, et dans la méthode get_example Vous pouvez voir que chaque image doit être lue et renvoyée avec l'étiquette correspondante.
2. Apprenez à utiliser la classe de jeu de données créée
Commençons maintenant à apprendre en utilisant cette classe CIFAR10. Voyons à quel point l'augmentation des données est efficace en utilisant le même grand réseau que nous avons utilisé précédemment. À l'exception de la classe de l'ensemble de données, y compris la fonction train, c'est presque le même que le code utilisé précédemment. La seule différence est le nombre d'époques et le nom du répertoire de destination.
model = train(DeepCNN(10), max_epoch=100, train_dataset=CIFAR10Augmented(), test_dataset=CIFAR10Augmented(False))
Résultat de sortie
epoch main/loss main/accuracy validation/main/loss validation/main/accuracy elapsed_time
1 2.023 0.248981 1.75221 0.322353 18.4387
2 1.51639 0.43716 1.36708 0.512639 36.482
3 1.25354 0.554177 1.17713 0.586087 54.6892
4 1.05922 0.637804 0.971438 0.665904 72.9602
5 0.895339 0.701886 0.918005 0.706409 91.4061
.
.
.
95 0.0877855 0.973171 0.726305 0.89162 1757.87
96 0.0780378 0.976012 0.943201 0.890725 1776.41
97 0.086231 0.973765 0.57783 0.890227 1794.99
98 0.0869593 0.973512 1.65576 0.878981 1813.52
99 0.0870466 0.972931 0.718033 0.891421 1831.99
100 0.079011 0.975332 0.754114 0.892815 1850.46
(Le journal étant long, le milieu est omis.)
Il a été constaté que la précision, qui avait culminé à environ 87% sans augmentation des données, peut être améliorée à 89% ou plus en appliquant une augmentation aux données d'entraînement. C'est une amélioration d'un peu plus de 2%.
Enfin, jetons un coup d'œil aux graphiques de perte et de précision.
Image(filename='DeepCNN_cifar10augmented_result/loss.png')
Résultat de sortie
Image(filename='DeepCNN_cifar10augmented_result/accuracy.png')
en conclusion
Dans cet article, à propos de Chainer
- Comment écrire une boucle d'apprentissage sans utiliser Trainer
- Comment utiliser Trainer
- Comment écrire votre propre modèle --Comment écrire votre propre classe de jeu de données
A été brièvement présenté. Je ne sais pas si cela sera fait dans un format pratique à l'avenir, mais j'aimerais écrire un commentaire sur quelque chose comme ce qui suit.
- Comment créer votre propre Updater et Itérateur qui composent le Trainer-
--Comment affiner les modèles pré-entraînés tels que
VGG16LayersetResNet50Layerssous le modulechainer.links.models.visionpour des tâches spécifiques --Comment faire l'extension
Nous souhaitons également la bienvenue à toute personne engagée dans le développement de Chainer! Chainer est un logiciel open source, nous allons donc évoluer en vous proposant les fonctions que vous souhaitez et en envoyant des pull requests. Si vous êtes intéressé, veuillez lire ce Guide de Contoribution, puis faites un numéro ou envoyez un PR. regarde s'il te plait. Nous vous attendons.
pfent/chainer https://github.com/pfnet/chainer
note de bas de page
[^ Données d'entraînement et de validation]: cet article se concentre sur l'explication de l'utilisation de Chainer, il ne fait donc pas de distinction claire entre les ensembles de données de validation et de test. Mais en réalité, il faut les distinguer. Normalement, vous supprimez certaines des données de formation de l'ensemble de données de formation et configurez l'ensemble de données de validation avec les données supprimées. Après cela, la procédure générale consiste d'abord à évaluer le modèle formé avec les données de formation avec les données de validation, puis à améliorer le modèle pour améliorer les performances avec les données de validation. Les données de test ne sont utilisées que pour évaluer les performances du modèle final (par exemple, à des fins de comparaison avec d'autres modèles) une fois tous les efforts terminés. Dans certains cas, plusieurs configurations de données d'apprentissage / de validation peuvent être préparées dans le but d'éviter le surajustement dû à l'amélioration du modèle à l'aide de données biaisées. [^ NN]: La précision des prévisions pour les données d'entraînement est celle des données si elles peuvent être trouvées en interrogeant certaines données extraites des données d'entraînement et en effectuant une recherche dans l'ensemble de données d'entraînement qui les contient. En répondant à l'étiquette ci-jointe, ce sera 100%.
Recommended Posts