Débarrassez-vous des images DICOM en Python
Pour faire simple, une image DICOM est une image qui est manipulée sur le protocole de communication (DICOM) pour échanger des images médicales entre appareils, et diverses informations complémentaires (informations patient, etc.) à l'image. C'est le type d'appareil, les informations de l'image elle-même et diverses métadonnées).
Lorsqu'un médecin effectue un examen tel qu'un scanner ou une IRM, certains hôpitaux peuvent vous demander d'utiliser l'image comme données. Si le format à ce moment est l'extension .dcm, il s'agit d'une image DICOM.
Se préparer à travailler avec DICOM en Python
Si vous souhaitez travailler avec des images DICOM en Python, il semble que vous utiliserez pydicom.
% anaconda show conda-forge/pydicom
Using Anaconda API: https://api.anaconda.org
Name: pydicom
Summary: Pure python package for DICOM medical file reading and writing
Access: public
Package Types: conda
Versions:
+ 0.9.9
To install this package with conda run:
conda install --channel https://conda.anaconda.org/conda-forge pydicom
C'est pourquoi conda install -c https://conda.anaconda.org/conda-forge pydicom` ``.
Préparez un échantillon d'image DICOM
Si vous n'avez malheureusement pas votre propre image IRM, téléchargez un exemple d'image. Il y a DICOM Image Library sur le site d'Osirix, qui est célèbre comme un visualiseur DICOM, alors procurez-vous-en un qui vous convient ici. Pour le moment, j'ai téléchargé une image IRM d'une tumeur cérébrale appelée BRAINIX. Lorsque ce fichier .zip est développé, il y a des images (accentuation T1, etc.) qui ont été soumises à divers traitements d'image pour être visualisées par l'interpréteur d'images après imagerie par IRM. Parmi elles, "T1-3D-FFE-C" J'ai décidé d'utiliser 100 fichiers dans le dossier "\ - \ 801 /".
Lire l'image DICOM
Vous trouverez ci-dessous un exemple de chargement d'une image à l'aide de la fonction pydicom.
Lire une image DICOM et consulter les métadonnées
Chargez l'image et affichez l'en-tête DICOM.
import dicom
d = dicom.read_file('BRAINIX/IM-0001-0075.dcm')
print(d)
En passant, si vous souhaitez afficher des métadonnées spécifiques, procédez comme suit.
print(d.Manufacturer)
Apparemment, le fabricant de l'équipement est "Philips Medical Systems".
print(d[0x0018, 0x0050])
Apparemment, l'épaisseur de la tranche est de 1,5 mm.
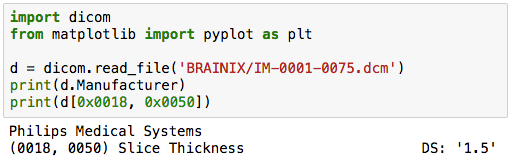
De cette manière, les métadonnées DICOM ont un nom de clé appelé balise et les clés sont numérotées. Et avec pydicom, vous pouvez afficher soit le nom de la clé, soit le numéro.
Lire une image DICOM et afficher l'image
Étant donné que la partie image réelle de l'image DICOM peut être extraite au format tableau de numpy, elle peut être affichée en utilisant soit opencv, soit matplotlib.

Lit plusieurs images DICOM et les convertit en une seule image tomographique continue
Soit dit en passant, l'image BRAINIX utilisée cette fois est une série de 100 images d'une épaisseur de 1,5 mm. Ces 100 images peuvent être lues et importées dans un seul tableau 3D.
import dicom
from matplotlib import pyplot as plt
%matplotlib inline
root_dir = './BRAINIX'
dcms = []
for d, s, fl in os.walk(root_dir):
for fn in fl:
if ".dcm" in fn.lower():
dcms.append(os.path.join(d, fn))
ref_dicom = dicom.read_file(dcms[0])
d_array = np.zeros((ref_dicom.Rows, ref_dicom.Columns, len(dcms)), dtype=ref_dicom.pixel_array.dtype)
for dcm in dcms:
d = dicom.read_file(dcm)
d_array[:, :, dcms.index(dcm)] = d.pixel_array
En bref, tous les fichiers du dossier BRAINIX avec l'extension .dcm sont lus et le contenu est copié dans un tableau tridimensionnel appelé d_array.
Si vous voulez afficher une tranche, par exemple la 50ème tranche, faites simplement pyplt.imshow (d_array [:,:, 49]) ``
Maintenant qu'il a été lu dans un tableau tridimensionnel, il est possible d'afficher trois sections.
import os
import dicom
from matplotlib import pyplot as plt
%matplotlib inline
#Lisez le fichier dcm dans le dossier BRAINIX
root_dir = './BRAINIX'
dcms = []
for d, s, fl in os.walk(root_dir):
for fn in fl:
if ".dcm" in fn.lower():
dcms.append(os.path.join(d, fn))
ref_dicom = dicom.read_file(dcms[0])
d_array = np.zeros((ref_dicom.Rows, ref_dicom.Columns, len(dcms)), dtype=ref_dicom.pixel_array.dtype)
for dcm in dcms:
d = dicom.read_file(dcm)
d_array[:, :, dcms.index(dcm)] = d.pixel_array
#3 Affichage en coupe
print(d_array.shape)
plt.subplot(1, 3, 1)
plt.imshow(d_array[127, :, :])
plt.subplot(1, 3, 2)
plt.imshow(d_array[:, 127, :])
plt.subplot(1, 3, 3)
plt.imshow(d_array[:, :, 49])

Traitez et enregistrez les images DICOM
Si vous souhaitez traiter l'image DICOM lue et l'enregistrer en écrasant l'image DICOM d'origine, vous devez modifier le tableau de pixel_array.
Cependant, l'image d'origine ne change pas simplement en éditant le tableau de pixel_array. Cela est dû au fait que les données d'image réelles de l'image DICOM sont stockées dans un tableau appelé PixelData et que l'interface de lecture est pixel_array. Par conséquent, si le contenu du pixel_array modifié est enregistré dans un tableau appelé img, par exemple, il est nécessaire de réécrire le contenu dans PixelData.
d = dicom.read_file(dcm)
img = d.pixel_array
#Traitement de l'img
d.PixelData = img.tostring()
d.save_as(dcm)
Essayez de recadrer l'image DICOM en cercle
Je ne sais pas pourquoi, mais je vais essayer de "découper l'image DICOM uniquement dans la partie centrale avec un cercle d'un diamètre égal à la moitié de la longueur et de la largeur de l'image".
Le code suivant crée une image avec 1 à l'intérieur et 0 à l'extérieur d'un cercle avec la moitié du diamètre de l'image. Si le rayon est r, la taille verticale et horizontale de l'image peut être obtenue à partir de ref_dicom.pixel_array.shape, définissez donc 1/4 de cette taille.
r = ref_dicom.pixel_array.shape[0] / 4
x, y = np.indices((ref_dicom.Rows, ref_dicom.Columns))
circle = (x - (ref_dicom.Columns / 2))**2 + (y - (ref_dicom.Rows / 2))**2 < r**2
mask = circle.astype(int)
En multipliant ce masque, l'intérieur du cercle est 1 et l'extérieur est 0, donc il peut être découpé en cercle.
###Lisez plusieurs fichiers image DICOM et découpez la partie centrale en cercle
import os
import dicom
from matplotlib import pyplot as plt
%matplotlib inline
#Lisez le fichier dcm dans le dossier BRAINIX
root_dir = './BRAINIX'
dcms = []
for d, s, fl in os.walk(root_dir):
for fn in fl:
if ".dcm" in fn.lower():
dcms.append(os.path.join(d, fn))
ref_dicom = dicom.read_file(dcms[0])
#Créez un masque à découper en cercle
r = ref_dicom.pixel_array.shape[0] / 4
x, y = np.indices((ref_dicom.Rows, ref_dicom.Columns))
circle = (x - (ref_dicom.Columns / 2))**2 + (y - (ref_dicom.Rows / 2))**2 < r**2
mask = circle.astype(int)
#Effectuer un traitement de masque sur toutes les images
d_array = np.zeros((ref_dicom.Rows, ref_dicom.Columns, len(dcms)), dtype=ref_dicom.pixel_array.dtype)
for dcm in dcms:
d = dicom.read_file(dcm)
img = d.pixel_array * mask
d_array[:, :, dcms.index(dcm)] = img
#3 Affichage en coupe
plt.figure(figsize=(8, 6))
plt.subplot(1, 3, 1)
plt.imshow(d_array[127, :, :])
plt.subplot(1, 3, 2)
plt.imshow(d_array[:, 127, :])
plt.subplot(1, 3, 3)
plt.imshow(d_array[:, :, 49])
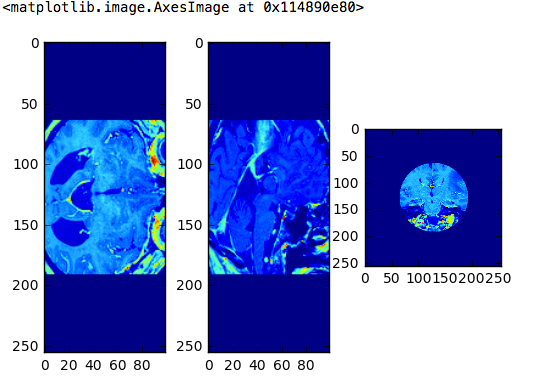
Lorsque vous traitez avec des images DICOM en Python, cela ressemble à ceci.
Code du jour
- https://github.com/fukuit/Python_SelfLearning/tree/master/dicom
Recommended Posts