Wie man IGV mit Socket-Kommunikation bedient und wie man einen Ruby Gem mit dieser Methode herstellt
Einführung
IGV ist eine Software namens Genome Browser. Sie können verschiedene Dateien durchsuchen, z. B. die BAM-Datei des ausgerichteten Lesevorgangs, die Bettdatei, in der die Positionsinformationen zum Genom kommentiert sind, und die GFF3-Datei, in der die genetischen Informationen geschrieben sind. Wenn Sie sich mit Bioinformatik befasst haben, kennen Sie diese Software wahrscheinlich.
- Das Bild stammt von IGV-Homepage
Obwohl es sich um ein solches IGV handelt, kann es einige Zeit dauern, es zu bedienen, während Sie mit der Maus klicken.
- Geben Sie das Referenzgenom an
- Laden Sie die erforderlichen Dateien
- Schauen Sie sich nacheinander an den Orten um, auf die Sie achten
- Machen Sie einen Screenshot und speichern Sie ihn als Bild
Es macht Spaß, die Ergebnisse visuell zu überprüfen, und ich denke, es ist eine sehr wichtige Aufgabe, aber andererseits möchte ich auch Routineaufgaben so weit wie möglich automatisieren. Erwägen Sie daher, das IGV über eine Programmiersprache zu betreiben.
- In letzter Zeit scheint igv.js, das mit JavaScript erstellt wurde, aktiv entwickelt und weit verbreitet worden zu sein. .. Dies wird in diesem Artikel aufgrund unzureichender Forschung nicht behandelt. (Aber irgendwann möchte ich einen Qiita-Artikel machen.)
Betreiben Sie IGV über Socket-Kommunikation
Tatsächlich kann IGV über die Socket-Kommunikation von Port 60151 aus betrieben werden.
In der Menüleiste befindet sich unter Ansicht> Einstellungen eine Registerkarte mit dem Namen Erweitert. Wenn Port aktivieren nicht aktiviert ist, überprüfen Sie hier.
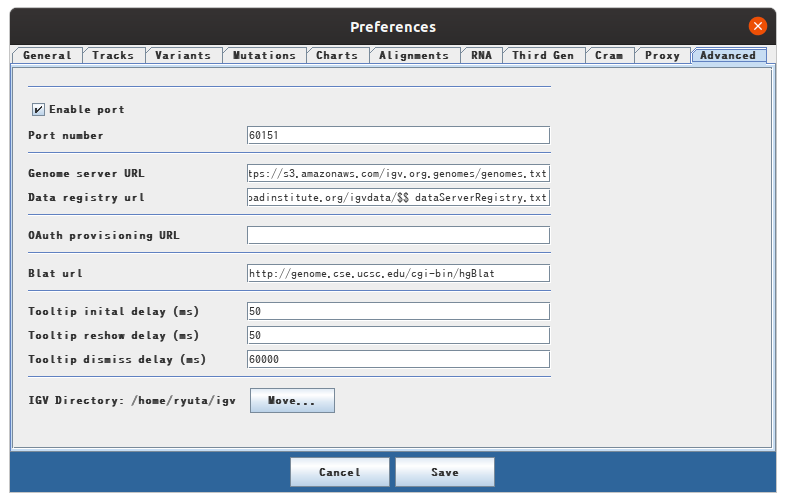
Sie können dann Port 60151 verwenden, um das IGV mithilfe der Socket-Kommunikation zu steuern.
Liste der Befehle, mit denen IGV betrieben werden kann
Ich habe die offizielle Referenz mit DeepL-Übersetzung ins Japanische übersetzt.
| Command | Description |
|---|---|
| new | Erstellen Sie eine neue Sitzung. Entfernen Sie alle Spuren mit Ausnahme der Standard-Genom-Annotation. |
| load file | Lesen Sie Daten oder Sitzungsdateien. Geben Sie den vollständigen Pfad oder die URL an, die durch Kommas getrennt sind. |
| collapse trackName | Reduziert den angegebenen Tracknamen. Wenn Sie trackName nicht angeben, werden alle Tracks reduziert. |
| echo | In Beantwortung"echo "Kehrt zurück.(zum Test) |
| exit | Beenden Sie die IGV-Anwendung. |
| expand trackName | Erweitert den angegebenen Tracknamen. Wenn trackName nicht angegeben ist, werden alle Tracks erweitert. |
| genome genomeIdOrPath | Wählen Sie ein Genom anhand der ID aus oder laden Sie das Genom (indiziertes Fasta) aus dem angegebenen Pfad. |
| goto locus or listOfLoci | Scrollen Sie zu einem einzelnen Ort oder einer durch Leerzeichen getrennten Liste von Orten. Wenn eine Liste bereitgestellt wird, werden diese Sitzpositionen in einer geteilten Bildschirmansicht angezeigt. Jede im IGV-Suchfeld gültige Syntax ist in Ordnung. |
| goto all | Scrollen Sie zur Ansicht des gesamten Genoms. |
| group option | Nur Ausrichtungsspur. Gruppenausrichtungen mit einer der folgenden Optionen: STRAND, SAMPLE, READ_GROUP, LIBRARY, FIRST_OF_PAIR_STRAND, TAG, PAIR_ORIENTATION, MATE_CHROMOSOME, SUPPLEMENTARY, MOVIE, ZMW, HAPLOTYPE, READ_ORDER, NONE, BASE_AT_POS |
| region chr start end | Definieren Sie eine Region von Interesse, die von zwei Loci umgeben ist (z. B. Region chr1 100 200). |
| maxPanelHeight height | Legt die Anzahl der vertikalen Pixel (Höhe) für jedes im Bild enthaltene Bedienfeld fest. Bilder, die mit Portbefehlen oder Batch-Skripten erstellt wurden, sind nicht auf die auf dem Bildschirm angezeigten Daten beschränkt. Mit anderen Worten, Sie können das gesamte Bedienfeld in Ihr Bild aufnehmen, nicht nur das, was im scrollbaren Bildschirmbereich angezeigt wird. Der Standardwert für diese Einstellung ist 1000. Erhöhen Sie diesen Wert, um mehr Daten anzuzeigen, oder verringern Sie diesen Wert, um kleinere Bilder zu erstellen. |
| setLogScale(true or false) | |
| setSleepInterval ms | Legt die Verzögerungszeit (Ruhezeit) in Millisekunden fest. Das Schlafintervall wird zwischen aufeinanderfolgenden Befehlen aufgerufen. |
| snapshotDirectory path | Legen Sie das Verzeichnis fest, in das das Bild geschrieben werden soll. |
| snapshot filename | Speichern Sie einen Schnappschuss des IGV-Fensters in einer Bilddatei. Wenn der Dateiname weggelassen wird, wird eine PNG-Datei mit dem Dateinamen geschrieben, der basierend auf der Flugbahn generiert wurde. Wenn Dateiname angegeben wird, bestimmt die Dateinamenerweiterung das Format der Bilddatei..png、.jpg oder.Muss svg sein. |
| sort option locus | Sortieren Sie Titel mit ausgerichteten oder segmentierten Kopienummern. Der Wert, der auf die Option für die segmentierte Kopiennummer angewendet wird, lautet(1)VERSTÄRKUNG UND LÖSCHUNG segmentierter Kopienummern,(2)Die Ausrichtungsspuren POSITION, STRAND, BASE, QUALITY, SAMPLE, READGROUP, INSERSTSIZE, FIRSTOFPAIRSTRAND, MATECHR, READORDER und READNAME. Bei dieser Option wird die Groß- und Kleinschreibung nicht berücksichtigt. Sie können den Ort angeben, um eine einzelne Position oder einen einzelnen Bereich zu definieren. Wenn Sie keine Option angeben, wird die Sortierung basierend auf dem angezeigten Bereich oder der Mittelposition des angezeigten Bereichs durchgeführt. |
| squish trackName | Squish den angegebenen trackName. trackName ist optional, andernfalls werden alle Anmerkungsspuren gequetscht. |
| viewaspairs trackName | Ausrichtungsspur-Anzeigemodus"View as pairs "Einstellen. |
| preference key value | Setzt die Einstellung mit dem Namen key vorübergehend auf den angegebenen Wert. Diese Einstellung ist nur gültig, bis das IGV heruntergefahren wird. |
Manipulieren Sie IGV über die Programmiersprache
Java
Da IGV eine in Java entwickelte Software ist, werden offizielle Beispiele auch in Java geschrieben.
Socket socket = new Socket("127.0.0.1", 60151);
PrintWriter out = new PrintWriter(socket.getOutputStream(), true);
BufferedReader in = new BufferedReader(new InputStreamReader(socket.getInputStream()));
out.println("load na12788.bam,n12788.tdf");
String response = in.readLine();
System.out.println(response);
out.println("genome hg18");
response = in.readLine();
System.out.println(response);
out.println("goto chr1:65,827,301");
//out.println("goto chr1:65,839,697");
response = in.readLine();
System.out.println(response);
out.println("snapshotDirectory /screenshots");
response = in.readLine();
System.out.println(response);
out.println("snapshot");
response = in.readLine();
System.out.println(response);
R
Was ist nun mit anderen Programmiersprachen als Java? Wenn Sie R verwenden, bin ich nicht sehr vertraut damit, aber Sie sollten es verwenden, da eine Bibliothek für den Betrieb von IGV aus der Umgebung von Bioconductor bereitgestellt wird. Eine kleine Suche trifft die Software igvR. Dies scheint igv.js zu verwenden, sodass das IGV möglicherweise nicht automatisch auf dem Desktop betrieben wird ...
Python
Es ist ein etwas altes Skript, aber igv.py wurde von Brent Pedersen erstellt, der kürzlich energisch Bioinformatik-Tools mit der Nim-Sprache entwickelt hat. -Es gibt ein Tool namens playground / blob / master / igv / igv.py). Dies ist eine kleine Bibliothek, die die Socket-Kommunikation oben umschließt.
Ruby
Ich mag Ruby. ** Nani Nani? Gibt es nur wenige Werkzeuge? Dann kannst du es selbst machen! ** Unter Bezugnahme auf das obige Skript von Brent Pedersen habe ich ein Tool namens ruby-igv erstellt, mit dem IGV in der Ruby-Sprache ausgeführt werden kann. Jetzt können Sie IGV problemlos über die Ruby-Sprache bedienen.
https://github.com/kojix2/ruby-igv
Die Verwendung ist wie folgt.
igv = IGV.new
igv.load 'na12788.bam'
igv.genome 'hg18'
igv.go 'goto chr1:65,827,301'
igv.save 'image.png'
Es ist immer noch ein frisch gemachtes Werkzeug. Wenn Sie auf Ecken und Kanten stoßen, Fehler finden oder Anfragen finden, melden Sie diese bitte an issue auf Github. Pull-Anfragen sind natürlich auch willkommen.
abschließend
Die Aufmerksamkeit beschränkt sich nicht nur auf die Bioinformatik, sondern auch darauf, wie vorhandene Tools kombiniert werden können, um den Zweck zu erreichen, und wie die Tools beherrscht werden. Und wenn Sie ein Werkzeug herstellen, denken Sie möglicherweise, dass Zweck und Mittel umgekehrt sind. Aber in gewissem Sinne denke ich, dass es eine sehr egoistische und engstirnige Denkweise ist. Je mehr Menschen mehr Tools erstellen und veröffentlichen, desto bequemer, expandierender und erfolgreicher wird die Welt. Lassen Sie uns Tools freier erstellen und veröffentlichen. (Weil es nicht Ruby sein muss)
