[Apprentissage automatique] Résumé et exécution de l'évaluation / des indicateurs du modèle (avec jeu de données Titanic)
J'ai rédigé un résumé de Cross Validation, de détermination d'hyperparamètres, de courbe ROC, AUC, etc., et une démonstration d'exécution en Python en relation avec le sobre mais important «évaluation / index du modèle».
Cet article est le 7ème jour du Calendrier de l'Avent Qiita Machine Learning 2015. Quand je l'ai regardé, 12/7 était gratuit, alors je l'ai écrit à la hâte: sourire:
Le code complet peut être trouvé dans le référentiel GitHub ici [https://github.com/matsuken92/Qiita_Contents/blob/master/Model_evaluation/Model_evaluation.ipynb).
0. Ensemble de données "Titanic"
Utilisez le jeu de données Titanic familier. Données sur les survivants du navire à passagers Titanic, souvent utilisées comme données de démonstration pour la classification.
Tout d'abord, le prétraitement et l'importation de données
J'ai un ensemble de données dans seaborn, donc je vais l'utiliser.
%matplotlib inline
import numpy as np
import pandas as pd
from time import time
from operator import itemgetter
import matplotlib as mpl
import matplotlib.pyplot as plt
from tabulate import tabulate
import seaborn as sns
sns.set(style="whitegrid", color_codes=True)
from sklearn import cross_validation
from sklearn import datasets
from sklearn import svm
from sklearn.ensemble import RandomForestClassifier
from sklearn.cross_validation import cross_val_score
from sklearn import grid_search
from sklearn.cross_validation import KFold
from sklearn.cross_validation import StratifiedKFold
from sklearn.metrics import classification_report, roc_auc_score, precision_recall_curve, auc, roc_curve
titanic = sns.load_dataset("titanic")
Ce sont les données. Les 5 premières lignes s'affichent.
headers = [c for c in titanic.columns]
headers.insert(0,"ID")
print tabulate(titanic[0:5], headers, tablefmt="pipe")
| ID | survived | pclass | sex | age | sibsp | parch | fare | embarked | class | who | adult_male | deck | embark_town | alive | alone |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | 3 | male | 22 | 1 | 0 | 7.25 | S | Third | man | 1 | nan | Southampton | no | 0 |
| 1 | 1 | 1 | female | 38 | 1 | 0 | 71.2833 | C | First | woman | 0 | C | Cherbourg | yes | 0 |
| 2 | 1 | 3 | female | 26 | 0 | 0 | 7.925 | S | Third | woman | 0 | nan | Southampton | yes | 1 |
| 3 | 1 | 1 | female | 35 | 1 | 0 | 53.1 | S | First | woman | 0 | C | Southampton | yes | 0 |
| 4 | 0 | 3 | male | 35 | 0 | 0 | 8.05 | S | Third | man | 1 | nan | Southampton | no | 1 |
#Faire des variables catégoriques des variables factices
def convert_dummies(df, key):
dum = pd.get_dummies(df[key])
ks = dum.keys()
print "Removing {} from {}...".format(ks[0], key)
dum.columns = [key + "_" + str(k) for k in ks]
df = pd.concat((df, dum.ix[:,1:]), axis=1)
df = df.drop(key, axis=1)
return df
titanic = convert_dummies(titanic, "who")
titanic = convert_dummies(titanic, "class")
titanic = convert_dummies(titanic, "sex")
titanic = convert_dummies(titanic, "alone")
titanic = convert_dummies(titanic, "embark_town")
titanic = convert_dummies(titanic, "deck")
titanic = convert_dummies(titanic, "embarked")
titanic['age'] = titanic.age.fillna(titanic.age.median())
titanic['adult_male'] = titanic.adult_male.map( {True: 1, False: 0} ).astype(int)
titanic['alone'] = titanic.adult_male.map( {True: 1, False: 0} ).astype(int)
#Supprimer les variables inutilisées
titanic = titanic.drop("alive", axis=1)
titanic = titanic.drop("pclass", axis=1)
1. Division de l'ensemble de données
1-1. Méthode d'exclusion
Un certain pourcentage des données sur les enseignants dont nous disposons actuellement est divisé en «données de formation» et «données de test» pour l'apprentissage et l'évaluation. Par exemple, si le rapport entre les données d'entraînement et les données de test est de 80:20, cela ressemble à ceci.

#Données d'entraînement(80%),données de test(20%)Diviser en
target = titanic.ix[:, 0]
data = titanic.ix[:, [1,2,3,4,5,6,7,8,9,10,11,12,14]]
X_train, X_test, y_train, y_test = cross_validation.train_test_split(data, target, test_size=0.2, random_state=None)
print [d.shape for d in [X_train, X_test, y_train, y_test]]
out
[(712, 23), (179, 23), (712,), (179,)]
- L'augmentation de la quantité de données d'entraînement augmente la précision de l'apprentissage, mais diminue la précision de l'évaluation du modèle.
- L'augmentation de la quantité de données de test augmente la précision de l'évaluation du modèle mais diminue la précision de l'apprentissage.
Faites attention au compromis et décidez du ratio.
# SVM(Noyau linéaire)Classer et calculer le taux d'erreur
clf = svm.SVC(kernel='linear', C=1).fit(X_train, y_train)
print u"Taux d'erreur de réaffectation:", 1 - clf.score(X_train, y_train)
print u"Taux d'erreur de maintien:", 1 - clf.score(X_test, y_test)
out
Taux d'erreur de réaffectation: 0.162921348315
Taux d'erreur de tenue: 0.212290502793
# SVM(noyau rbf)Classer et calculer le taux d'erreur
clf = svm.SVC(kernel='rbf', C=1).fit(X_train, y_train)
print u"Taux d'erreur de réaffectation:", 1 - clf.score(X_train, y_train)
print u"Taux d'erreur de maintien:", 1 - clf.score(X_test, y_test)
out
Taux d'erreur de réaffectation: 0.101123595506
Taux d'erreur de tenue: 0.268156424581
1-2. Validation croisée (CV): pli k stratifié
K-fold Tout d'abord, à propos du simple pli en K. Prenons le cas où il y a 30 données. n_folds est le nombre de divisions, divise l'ensemble de données que vous avez maintenant par le nombre de divisions et génère toutes les combinaisons sous la forme d'une liste d'indices comme indiqué ci-dessous, qui sont les données de test.

# KFold
# n_Divisez les données par la valeur numérique spécifiée par des plis. n_folds=S'il est 5, il est divisé en 5
#Ensuite, l'un d'eux est utilisé comme données de test et cinq modèles sont générés.
kf = KFold(30, n_folds=5,shuffle=False)
for tr, ts in kf:
print("%s %s" % (tr, ts))
out
[ 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29] [0 1 2 3 4 5]
[ 0 1 2 3 4 5 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29] [ 6 7 8 9 10 11]
[ 0 1 2 3 4 5 6 7 8 9 10 11 18 19 20 21 22 23 24 25 26 27 28 29] [12 13 14 15 16 17]
[ 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 24 25 26 27 28 29] [18 19 20 21 22 23]
[ 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23] [24 25 26 27 28 29]
Stratified k-fold
À partir du facteur K ci-dessus, une méthode de division de l'ensemble de données de manière à conserver chaque rapport de classe dans l'ensemble de données à portée de main. Le cross_validation.cross_val_score utilisé dans la section suivante l'adopte.
# StratifiedKFold
#Une version améliorée de KFold qui fait correspondre le taux d'extraction de chaque classe au rapport des données d'origine
label = np.r_[np.repeat(0,20), np.repeat(1,10)]
skf = StratifiedKFold(label, n_folds=5, shuffle=False)
for tr, ts in skf:
print("%s %s" % (tr, ts))
out
[ 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 22 23 24 25 26 27 28 29] [ 0 1 2 3 20 21]
[ 0 1 2 3 8 9 10 11 12 13 14 15 16 17 18 19 20 21 24 25 26 27 28 29] [ 4 5 6 7 22 23]
[ 0 1 2 3 4 5 6 7 12 13 14 15 16 17 18 19 20 21 22 23 26 27 28 29] [ 8 9 10 11 24 25]
[ 0 1 2 3 4 5 6 7 8 9 10 11 16 17 18 19 20 21 22 23 24 25 28 29] [12 13 14 15 26 27]
[ 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 20 21 22 23 24 25 26 27] [16 17 18 19 28 29]
Essayez de courir
# SVM(Noyau linéaire)Classer et calculer le taux d'erreur
#Calculez chaque score avec un pli en K stratifié divisé en 5
clf = svm.SVC(kernel='rbf', C=1)
scores = cross_validation.cross_val_score(clf, data, target, cv=5,)
print "scores: ", scores
print("Accuracy: %0.2f (+/- %0.2f)" % (scores.mean(), scores.std() * 2))
Puisque 5 est spécifié pour n_folds, 5 scores différents sont affichés. La précision est la valeur moyenne et l'écart type.
out
scores: [ 0.67039106 0.70949721 0.74157303 0.74719101 0.78531073]
Accuracy: 0.73 (+/- 0.08)
2. Comment trouver de meilleurs hyper paramètres
2-1. Exhaustive Grid Search En d'autres termes, c'est un moyen d'essayer tous les hyperparamètres que vous avez définis à partir de zéro pour savoir lequel est le meilleur. Cela prendra du temps, mais nous essaierons tout ce que vous spécifiez pour que vous en trouviez probablement un bon.
Tout d'abord, définissez les valeurs possibles des paramètres comme suit.
param_grid = [
{'kernel': ['rbf','linear'], 'C': np.linspace(0.1,2.0,20),}
]
Passez-le à grid_search.GridSearchCV avec SVC (Support Vector Classifier) et exécutez-le.
#Courir
svc = svm.SVC(random_state=None)
clf = grid_search.GridSearchCV(svc, param_grid)
res = clf.fit(X_train, y_train)
#Voir les résultats
print "score: ", clf.score(X_test, y_test)
print "best_params:", res.best_params_
print "best_estimator:", res.best_estimator_
Il essaie tous les paramètres spécifiés et affiche le score avec le meilleur résultat, quel paramètre était bon et le paramètre spécifié détaillé.
out
0.787709497207
{'kernel': 'linear', 'C': 0.40000000000000002}
SVC(C=0.40000000000000002, cache_size=200, class_weight=None, coef0=0.0,
degree=3, gamma=0.0, kernel='linear', max_iter=-1, probability=False,
random_state=None, shrinking=True, tol=0.001, verbose=False)
Cependant, cela a pris du temps après tout. Lorsque le nombre de paramètres que vous souhaitez essayer augmente, vous ne pourrez pas tous les voir. Ensuite, je vais choisir une méthode qui incorpore le caractère aléatoire dans la façon de sélectionner les paramètres.
2-2. Randomized Parameter Optimization
Essayez d'augmenter les paramètres que vous souhaitez essayer. Cette fois, nous utiliserons Random forest, qui a de nombreux paramètres.
param_dist = {'n_estimators': range(4,20,2), 'min_samples_split': range(1,30,2), 'criterion':['gini','entropy']}
Exécutons-le et voyons combien de temps le temps de traitement est. (* Si le nombre de combinaisons de paramètres est inférieur ou égal à 20, une erreur apparaîtra indiquant "Utiliser la recherche par grille")
n_iter_search = 20
rfc = RandomForestClassifier(max_depth=None, min_samples_split=1, random_state=None)
random_search = grid_search.RandomizedSearchCV(rfc,
param_distributions=param_dist,
n_iter=n_iter_search)
start = time()
random_search.fit(X_train, y_train)
end = time()
print"Nombre de paramètres: {0},temps écoulé: {1:0.3f}Secondes".format(n_iter_search, end - start)
out
Nombre de paramètres: 20,temps écoulé: 0.805 secondes
Les 3 principaux réglages de paramètres sont indiqués ci-dessous.
#Top 3 des paramètres
top_scores = sorted(random_search.grid_scores_, key=itemgetter(1), reverse=True)[:3]
for i, score in enumerate(top_scores):
print("Model with rank: {0}".format(i + 1))
print("Mean validation score: {0:.3f} (std: {1:.3f})".format(
score.mean_validation_score,
np.std(score.cv_validation_scores)))
print("Parameters: {0}".format(score.parameters))
print("")
out
Model with rank: 1
Mean validation score: 0.834 (std: 0.007)
Parameters: {'min_samples_split': 7, 'n_estimators': 10, 'criterion': 'gini'}
Model with rank: 2
Mean validation score: 0.826 (std: 0.010)
Parameters: {'min_samples_split': 25, 'n_estimators': 18, 'criterion': 'entropy'}
Model with rank: 3
Mean validation score: 0.823 (std: 0.022)
Parameters: {'min_samples_split': 19, 'n_estimators': 12, 'criterion': 'gini'}
3. Indice d'évaluation
Considérez le résultat du diagnostic d'une certaine maladie comme exemple. À ce moment-là, il existe quatre résultats possibles, comme indiqué ci-dessous, en fonction du résultat du diagnostic et de la valeur réelle. Le tableau est le suivant.

De base,
- T(true), F(false)
- P(positive), N(negative)
En cas d'examen avec la combinaison de vrai signifie "le résultat du diagnostic et la valeur vraie correspondent", faux signifie "le résultat du diagnostic et la valeur vraie sont différents" Le positif est «malade», le négatif est «pas malade» Représente.
Sur cette base, les indices d'évaluation suivants sont calculés.
Taux de réponse correct [Précision]
Taux de précision [Précision]
Le pourcentage de réponses correctes parmi ceux dont le diagnostic est positif.
Taux de rappel [Rappel]
Taux de réponse correct parmi ceux dont la vraie valeur est mauvaise
Valeur F (mesure F)
Moyenne harmonisée de précision et de rappel.
3-1. Essayez de calculer
# SVM(Noyau linéaire)Classer et calculer l'indice d'évaluation
clf = svm.SVC(kernel='linear', C=1, probability=True).fit(X_train, y_train)
print u"Accuracy:", clf.score(X_test, y_test)
y_pred = clf.predict(X_test)
print classification_report(y_test, y_pred, target_names=["not Survived", "Survived"])
out
precision recall f1-score support
not Survived 0.80 0.86 0.83 107
Survived 0.77 0.68 0.72 72
avg / total 0.79 0.79 0.79 179
3-2. ROC et AUC
J'expliquerai l'index appelé AUC. Cela peut être dérivé de la courbe ROC (Receiver Operating Characteristic), mais dans l'article précédent,
[Statistiques] Comprenez ce qu'est la courbe ROC par animation.
Veuillez consulter ici pour des explications détaillées dans.
L'aire sous cette courbe ROC est AUC (Area Under the Curve). (C'est vrai ...: sweat_smile :)
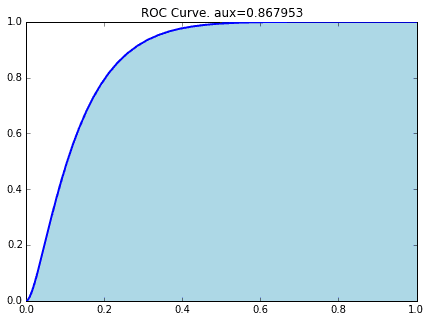
prob = clf.predict_proba(X_test)[:,1]
fpr, tpr, thresholds= roc_curve(y_test, prob)
plt.figure(figsize=(8,6))
plt.plot(fpr, tpr)
plt.title("ROC curve")
plt.xlabel("False Positive Rate")
plt.ylabel("True Positive Rate")
plt.show()
La courbe ROC lorsque les données Titanic sont classées par SVM (Linear Kernel) est la suivante.

#Calcul de l'ASC
precision, recall, thresholds = precision_recall_curve(y_test, prob)
area = auc(recall, precision)
print "Area Under Curve: {0:.3f}".format(area)
out
Area Under Curve: 0.800
référence
WEB Scikit Learn User Guide 3. Model selection and evaluation http://scikit-learn.org/stable/model_selection.html
** Livres ** "Première reconnaissance de formes" Yuzo Hirai "Introduction à l'apprentissage automatique pour le traitement du langage" Manabu Okumura
Recommended Posts