J'ai essayé de vérifier à quelle vitesse la mnist de l'exemple Chainer peut être accélérée en utilisant cython
Pourquoi faire
Il s'est accéléré depuis Chainer 1.5. Il a été mentionné que l'utilisation de cython était l'une des raisons de l'accélération, j'ai donc eu une question simple sur la vitesse à laquelle je réécrirais l'exemple avec cython, alors je l'ai essayé.
Spécifications PC
OS:OS X Yosemite CPU: 2.7GHz Intel Core i5 Memory:8GHz DDR3
conditions
Utiliser l'exemple Mnist Nombre d'apprentissage: 20 fois Les données ont été téléchargées à l'avance
Visualisation
Visualisation avec profiler
Utiliser pycallgraph
http://pycallgraph.slowchop.com
Installez graphviz
http://www.graphviz.org/Download_macos.php
Installez X11 (pour Yosemite)
http://www.xquartz.org/
Lorsque a échoué avec le code d'erreur 256 apparaît
https://github.com/gak/pycallgraph/issues/100
Lors de l'utilisation de pycallgraph
python pycallgraph graphviz -- ./nom de fichier.py
Ce que je veux faire
1: Visualisation et profilage du traitement normal 2: cython simple 3: réglage du type statique par cdef 4: Cythonalisation de modules externes
Etat initial
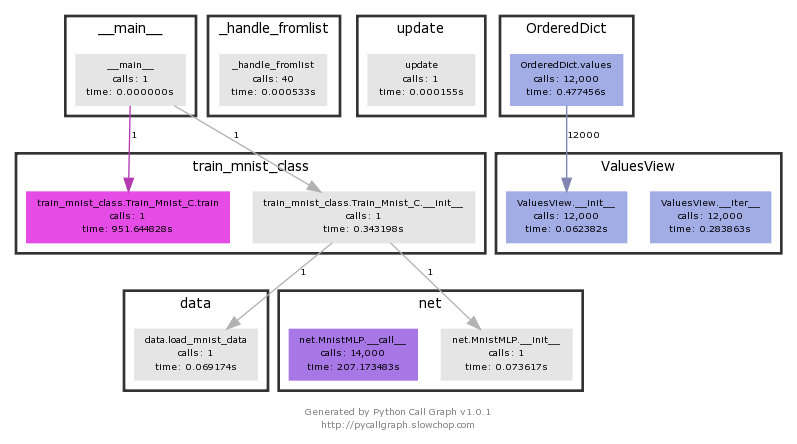
La visualisation vous donne une idée de ce qui prend du temps. train_mnist.Train_Mnist.train Vous pouvez voir que cela prend 951 secondes.
Le résultat d'un profil normal est ci-dessous.
ncalls: nombre d'appels tottime: temps total passé par cette fonction percall: temps total divisé par ncalls cumtime: Temps total passé (du début à la fin) de cette fonction, y compris les fonctions subordonnées. Cet élément est également mesuré avec précision dans les fonctions récursives. percall: cumtime divisé par le nombre d'appels primitifs
Cette fois, le temps de traitement est différent de ce qui précède car le code a été modifié pour la commodité de cython. Je voulais vraiment utiliser pycallgraph avec cython, mais je n'ai pas pu l'utiliser en raison de mon manque de connaissances. Si quelqu'un sait comment l'utiliser, veuillez me le faire savoir (le traitement de la partie cithon ne sera pas répertorié dans un usage normal)
Il se termine en 755,154 secondes.
Méthode d'exécution
python -m cProfile
Profile.prof
37494628 function calls (35068627 primitive calls) in 755.154 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 448.089 0.006 448.651 0.006 adam.py:27(update_one_cpu)
114000 187.057 0.002 187.057 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 31.576 0.000 31.576 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 23.122 0.002 163.601 0.014 variable.py:216(backward)
En vous basant sur la règle 2: 8, concentrez-vous sur la partie qui prend le plus de temps à traiter. Vous pouvez voir que adam.py prend presque la plupart du temps de traitement, et les opérations matricielles de numpy continuent de consacrer le temps de traitement.
cython
Je voulais aussi le représenter graphiquement dans cython, mais je n'avais pas assez de connaissances sur l'auteur et je ne pouvais pas représenter graphiquement uniquement la partie traitement de cython, alors je l'ai profilé.
Le résultat est plus lent que 800 secondes
Profile.prof
37466504 function calls (35040503 primitive calls) in 800.453 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 473.638 0.007 474.181 0.007 adam.py:27(update_one_cpu)
114000 199.589 0.002 199.589 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 33.706 0.000 33.706 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 24.754 0.002 173.816 0.014 variable.py:216(backward)
28000 9.944 0.000 10.392 0.000
La partie traitement de adam.py et variable.py est plus lente qu'avant la cythonisation. Il est possible qu'il soit lent en raison de la coopération entre le langage c converti par cython et le traitement externe de python.
cdef
J'ai défini cdef avec l'espoir qu'il serait plus rapide de définir un type statique à l'avance en utilisant cdef.
Préparation préalable
Quand je l'ai utilisé sur mac tel quel, une erreur s'est produite, j'ai donc pris diverses mesures.
Lorsque j'essaye d'utiliser cimport, j'obtiens l'erreur suivante.
/Users/smap2/.pyxbld/temp.macosx-10.10-x86_64-3.4/pyrex/train_mnist_c2.c:242:10: fatal error: 'numpy/arrayobject.h' file not found
Dans le répertoire suivant
/usr/local/include/
Copiez ou transmettez le répertoire d'en-tête trouvé par la commande suivante
find / -name arrayobject.h -print 2> /dev/null
C'était 776 secondes.
Profile.prof
37466756 function calls (35040748 primitive calls) in 776.901 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 458.284 0.006 458.812 0.006 adam.py:27(update_one_cpu)
114000 194.834 0.002 194.834 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 33.120 0.000 33.120 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 24.025 0.002 168.772 0.014 variable.py:216(backward)
C'est une amélioration par rapport à la cythonisation simple, mais comme il n'y a pas beaucoup de changement dans adam.py et variable.py, il est plus lent que le traitement python en raison du langage C supplémentaire et du traitement de conversion du langage Python.
cythonisation de adam.py
J'ai essayé d'accélérer la partie qui prend le plus de temps en convertissant adam.py en cython.
En conséquence, il a montré l'effet d'être environ 30 secondes plus rapide.
Profile.prof
37250749 function calls (34824741 primitive calls) in 727.414 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 430.495 0.006 430.537 0.006 optimizer.py:388(update_one)
114000 180.775 0.002 180.775 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 30.647 0.000 30.647 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 21.766 0.002 157.230 0.013 variable.py:216(backward)
Le temps de traitement de optimizer.py contenant adam.py est environ 20 secondes plus rapide que celui de python. Cela a fonctionné et est devenu plus rapide.
Résumé
Au lieu d'essayer simplement d'accélérer par cython, j'ai trouvé qu'il y a une possibilité que cela soit efficace si je le profile et que je ne cython que là où cela fonctionne vraiment. C'était un calendrier de l'Avent que j'ai pu découvrir que le cycle d'hypothèses, de visualisation et de vérification est important, plutôt que d'essayer de s'en débarrasser.
Le code que j'ai essayé d'exécuter est répertorié ci-dessous. https://github.com/SnowMasaya/chainer_cython_study
référence
Chainer: a neural network framework https://github.com/pfnet/chainer
Recommended Posts