Guide de l'utilisateur Pandas "Manipulation des données manquantes" (Document officiel de traduction en japonais)
Cet article est une traduction automatique partielle de la documentation officielle de Pandas Guide de l'utilisateur - Travailler avec des données manquantes. C'est une modification d'une phrase contre nature.
Si vous avez des erreurs de traduction, des traductions alternatives, des questions, etc., veuillez utiliser la section commentaires ou modifier la demande.
Manipuler les données manquantes
Cette section décrit les valeurs manquantes (NA) pour les pandas.
: information_source: ** Remarque ** J'ai choisi d'utiliser
NaNen interne pour indiquer les données manquantes, principalement pour des raisons de simplicité et de performances. À partir de pandas 1.0, certains types de données optionnels commencent à essayer des scalaires natifsNAen utilisant une approche basée sur un masque. Voir [here](# experimental na scalar pour indiquer les valeurs manquantes) pour plus d'informations.
Valeur considérée comme "manquante"
Étant donné que les données existent sous de nombreuses formes et formats, les pandas veulent être flexibles pour traiter les données manquantes. NaN est le marqueur de valeur manquante par défaut pour la vitesse de calcul et la commodité, mais il devrait être facile à détecter pour les types de données à virgule flottante, entier, booléen et divers pour les objets généraux. .. Cependant, dans de nombreux cas, «Aucun» de Python est également trouvé, et «manquant» ou «indisponible» ou «NA» doit être pris en compte.
: information_source: ** Remarque ** Si vous voulez que le calcul considère ʻinf
et-infcomme "NA", vous pouvez le définir avecpandas.options.mode.use_inf_as_na = True`.
In [**]: df = pd.DataFrame(np.random.randn(5, 3), index=['a', 'c', 'e', 'f', 'h'],
....: columns=['one', 'two', 'three'])
....:
In [**]: df['four'] = 'bar'
In [**]: df['five'] = df['one'] > 0
In [**]: df
Out[**]:
one two three four five
a 0.469112 -0.282863 -1.509059 bar True
c -1.135632 1.212112 -0.173215 bar False
e 0.119209 -1.044236 -0.861849 bar True
f -2.104569 -0.494929 1.071804 bar False
h 0.721555 -0.706771 -1.039575 bar True
In [**]: df2 = df.reindex(['a', 'b', 'c', 'd', 'e', 'f', 'g', 'h'])
In [**]: df2
Out[**]:
one two three four five
a 0.469112 -0.282863 -1.509059 bar True
b NaN NaN NaN NaN NaN
c -1.135632 1.212112 -0.173215 bar False
d NaN NaN NaN NaN NaN
e 0.119209 -1.044236 -0.861849 bar True
f -2.104569 -0.494929 1.071804 bar False
g NaN NaN NaN NaN NaN
h 0.721555 -0.706771 -1.039575 bar True
Pour faciliter la détection des valeurs manquantes (parfois via des tableaux de différents types de données), pandas isna () /pandas.isna.html#pandas.isna) </ code> et [notna ()](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.notna.html La fonction # pandas.notna) </ code> est fournie. Ce sont également des méthodes d'objets Series et DataFrame.
In [**]: df2['one']
Out[**]:
a 0.469112
b NaN
c -1.135632
d NaN
e 0.119209
f -2.104569
g NaN
h 0.721555
Name: one, dtype: float64
In [**]: pd.isna(df2['one'])
Out[**]:
a False
b True
c False
d True
e False
f False
g True
h False
Name: one, dtype: bool
In [**]: df2['four'].notna()
Out[**]:
a True
b False
c True
d False
e True
f True
g False
h True
Name: four, dtype: bool
In [**]: df2.isna()
Out[**]:
one two three four five
a False False False False False
b True True True True True
c False False False False False
d True True True True True
e False False False False False
f False False False False False
g True True True True True
h False False False False False
: avertissement: ** Avertissement ** Il faut noter qu'en Python (et NumPy), «nan» n'est pas équivalent et «None» est ** équivalent **. Notez que pandas / NumPy traite «None» comme «np.nan», tout en utilisant le fait que «np.nan! = Np.nan».
In [**]: None == None # noqa: E711 Out[**]: True In [**]: np.nan == np.nan Out[**]: FalsePar conséquent, par rapport à ce qui précède, les comparaisons d'équivalence scalaire pour «None / np.nan» ne fournissent pas d'informations utiles.
In [**]: df2['one'] == np.nan Out[**]: a False b False c False d False e False f False g False h False Name: one, dtype: bool
Type de données entier et données manquantes
Puisque NaN est un nombre à virgule flottante, une colonne d'entiers qui a au moins une valeur manquante sera convertie en un type de données de nombre à virgule flottante (pour plus d'informations, consultez Prise en charge des nombres entiers NA (https: // pandas. Voir pydata.org/pandas-docs/stable/user_guide/gotchas.html#gotchas-intna)). pandas fournit un tableau d'entiers qui peuvent contenir des valeurs manquantes. Cela peut être utilisé en spécifiant explicitement le type de données.
In [**]: pd.Series([1, 2, np.nan, 4], dtype=pd.Int64Dtype())
Out[**]:
0 1
1 2
2 <NA>
3 4
dtype: Int64
Il peut également être utilisé en spécifiant l'alias de chaîne «dtype = 'Int64» (notez le «I» supérieur »).
Voir Types de données entiers manquants (https://pandas.pydata.org/pandas-docs/stable/user_guide/integer_na.html#integer-na) pour plus d'informations.
Données de séries chronologiques (datetime)
Pour le type datetime64 \ [ns ], NaT représente la valeur manquante. Il s'agit d'une valeur sentinelle pseudo-native qui peut être représentée par un seul type de données NumPy (datetime64 \ [ns ]). L'objet pandas assure la compatibilité entre «NaT» et «NaN».
In [**]: df2 = df.copy()
In [**]: df2['timestamp'] = pd.Timestamp('20120101')
In [**]: df2
Out[**]:
one two three four five timestamp
a 0.469112 -0.282863 -1.509059 bar True 2012-01-01
c -1.135632 1.212112 -0.173215 bar False 2012-01-01
e 0.119209 -1.044236 -0.861849 bar True 2012-01-01
f -2.104569 -0.494929 1.071804 bar False 2012-01-01
h 0.721555 -0.706771 -1.039575 bar True 2012-01-01
In [**]: df2.loc[['a', 'c', 'h'], ['one', 'timestamp']] = np.nan
In [**]: df2
Out[**]:
one two three four five timestamp
a NaN -0.282863 -1.509059 bar True NaT
c NaN 1.212112 -0.173215 bar False NaT
e 0.119209 -1.044236 -0.861849 bar True 2012-01-01
f -2.104569 -0.494929 1.071804 bar False 2012-01-01
h NaN -0.706771 -1.039575 bar True NaT
In [**]: df2.dtypes.value_counts()
Out[**]:
float64 3
object 1
datetime64[ns] 1
bool 1
dtype: int64
Insérer les données manquantes
Vous pouvez insérer des valeurs manquantes simplement en les affectant à un conteneur. Les valeurs manquantes réelles utilisées sont sélectionnées en fonction du type de données.
Par exemple, un conteneur numérique utilise toujours «NaN», quel que soit le type de valeur manquante donné.
In [**]: s = pd.Series([1, 2, 3])
In [**]: s.loc[0] = None
In [**]: s
Out[**]:
0 NaN
1 2.0
2 3.0
dtype: float64
De même, les conteneurs de séries chronologiques utilisent toujours «NaT».
Pour les conteneurs d'objets, les pandas utiliseront la valeur donnée.
In [**]: s = pd.Series(["a", "b", "c"])
In [**]: s.loc[0] = None
In [**]: s.loc[1] = np.nan
In [**]: s
Out[**]:
0 None
1 NaN
2 c
dtype: object
Calcul des données manquantes
Les valeurs manquantes se propagent naturellement par des opérations arithmétiques entre les objets pandas.
In [**]: a
Out[**]:
one two
a NaN -0.282863
c NaN 1.212112
e 0.119209 -1.044236
f -2.104569 -0.494929
h -2.104569 -0.706771
In [**]: b
Out[**]:
one two three
a NaN -0.282863 -1.509059
c NaN 1.212112 -0.173215
e 0.119209 -1.044236 -0.861849
f -2.104569 -0.494929 1.071804
h NaN -0.706771 -1.039575
In [**]: a + b
Out[**]:
one three two
a NaN NaN -0.565727
c NaN NaN 2.424224
e 0.238417 NaN -2.088472
f -4.209138 NaN -0.989859
h NaN NaN -1.413542
Vue d'ensemble de la structure de données (et ici -docs / stable / reference / series.html # api-series-stats) et ici Toutes les statistiques descriptives et les méthodes de calcul décrites dans (Liste de) sont écrites pour expliquer les données manquantes. Par exemple:
- Lors de la somme des données, les valeurs NA (manquantes) sont traitées comme zéro. --Si toutes les données sont NA, le résultat sera 0.
cumsum () </ code> et < Méthodes cumulatives telles que code> cumprod () </ code> Ignore les valeurs NA par défaut, mais les conserve dans le tableau résultant. Pour remplacer ce comportement et inclure la valeur NA, utilisezskipna = False.
In [**]: df
Out[**]:
one two three
a NaN -0.282863 -1.509059
c NaN 1.212112 -0.173215
e 0.119209 -1.044236 -0.861849
f -2.104569 -0.494929 1.071804
h NaN -0.706771 -1.039575
In [**]: df['one'].sum()
Out[**]: -1.9853605075978744
In [**]: df.mean(1)
Out[**]:
a -0.895961
c 0.519449
e -0.595625
f -0.509232
h -0.873173
dtype: float64
In [**]: df.cumsum()
Out[**]:
one two three
a NaN -0.282863 -1.509059
c NaN 0.929249 -1.682273
e 0.119209 -0.114987 -2.544122
f -1.985361 -0.609917 -1.472318
h NaN -1.316688 -2.511893
In [**]: df.cumsum(skipna=False)
Out[**]:
one two three
a NaN -0.282863 -1.509059
c NaN 0.929249 -1.682273
e NaN -0.114987 -2.544122
f NaN -0.609917 -1.472318
h NaN -1.316688 -2.511893
Puissance totale / totale dans les données vides / manquantes
: avertissement: ** Avertissement ** Ce comportement est la norme actuelle dans la v0.22.0 et correspond à la valeur par défaut de
numpy. Auparavant, la puissance somme / somme pour tous les NA ou les Series / DataFrames vides renvoyait NaN. Pour plus d'informations, voir v0.22.0 whatsnew.
La somme des colonnes d'un Series ou DataFrame qui est vide ou tout NA est 0.
In [**]: pd.Series([np.nan]).sum()
Out[**]: 0.0
In [**]: pd.Series([], dtype="float64").sum()
Out[**]: 0.0
La puissance totale d'une série ou d'une colonne DataFrame qui est vide ou entièrement NA est 1.
In [**]: pd.Series([np.nan]).prod()
Out[**]: 1.0
In [**]: pd.Series([], dtype="float64").prod()
Out[**]: 1.0
Valeurs manquantes dans GroupBy
GroupBy exclut automatiquement les groupes NA. Ce comportement est conforme à R. Par exemple
In [**]: df
Out[**]:
one two three
a NaN -0.282863 -1.509059
c NaN 1.212112 -0.173215
e 0.119209 -1.044236 -0.861849
f -2.104569 -0.494929 1.071804
h NaN -0.706771 -1.039575
In [**]: df.groupby('one').mean()
Out[**]:
two three
one
-2.104569 -0.494929 1.071804
0.119209 -1.044236 -0.861849
Pour plus d'informations, consultez la section groupby ici [https://pandas.pydata.org/pandas-docs/stable/user_guide/groupby.html#groupby-missing).
Exclusion / remplir les données manquantes
L'objet pandas est équipé de diverses méthodes de manipulation de données pour traiter les données manquantes.
Remplissez les valeurs manquantes-fillna
fillna () </ code> est manquant Vous pouvez «remplir» les valeurs avec des données non manquantes.
** Remplacez les valeurs manquantes par des valeurs scalaires **
In [**]: df2
Out[**]:
one two three four five timestamp
a NaN -0.282863 -1.509059 bar True NaT
c NaN 1.212112 -0.173215 bar False NaT
e 0.119209 -1.044236 -0.861849 bar True 2012-01-01
f -2.104569 -0.494929 1.071804 bar False 2012-01-01
h NaN -0.706771 -1.039575 bar True NaT
In [**]: df2.fillna(0)
Out[**]:
one two three four five timestamp
a 0.000000 -0.282863 -1.509059 bar True 0
c 0.000000 1.212112 -0.173215 bar False 0
e 0.119209 -1.044236 -0.861849 bar True 2012-01-01 00:00:00
f -2.104569 -0.494929 1.071804 bar False 2012-01-01 00:00:00
h 0.000000 -0.706771 -1.039575 bar True 0
In [**]: df2['one'].fillna('missing')
Out[**]:
a missing
c missing
e 0.119209
f -2.10457
h missing
Name: one, dtype: object
** Remplissez les espaces avec des données avant ou arrière **
Propagez les valeurs non manquantes vers l'avant ou vers l'arrière en utilisant des paramètres de remplissage similaires à ceux de Reindex (https://pandas.pydata.org/pandas-docs/stable/user_guide/basics.html#basics-reindexing) Vous pouvez.
In [**]: df
Out[**]:
one two three
a NaN -0.282863 -1.509059
c NaN 1.212112 -0.173215
e 0.119209 -1.044236 -0.861849
f -2.104569 -0.494929 1.071804
h NaN -0.706771 -1.039575
In [**]: df.fillna(method='pad')
Out[**]:
one two three
a NaN -0.282863 -1.509059
c NaN 1.212112 -0.173215
e 0.119209 -1.044236 -0.861849
f -2.104569 -0.494929 1.071804
h -2.104569 -0.706771 -1.039575
** Limiter la quantité de remplissage **
Si vous souhaitez simplement combler des lacunes consécutives à un certain point de données, vous pouvez utiliser le mot-clé * limit *.
In [**]: df
Out[**]:
one two three
a NaN -0.282863 -1.509059
c NaN 1.212112 -0.173215
e NaN NaN NaN
f NaN NaN NaN
h NaN -0.706771 -1.039575
In [**]: df.fillna(method='pad', limit=1)
Out[**]:
one two three
a NaN -0.282863 -1.509059
c NaN 1.212112 -0.173215
e NaN 1.212112 -0.173215
f NaN NaN NaN
h NaN -0.706771 -1.039575
Les méthodes de remplissage disponibles sont:
| Méthode | mouvement |
|---|---|
| pad / ffill | Remplissez les trous vers l'avant |
| bfill / backfill | Remplissez les trous à l'envers |
L'utilisation de pad / ffill est si courante dans les données de séries chronologiques que la «dernière valeur connue» est disponible à tout moment.
ffill () </ code> est fillna Équivalent à (method = 'ffill') , [bfill ()](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.DataFrame.bfill.html# pandas.DataFrame.bfill) </ code> équivaut à fillna (method = 'bfill').
Remplissez les blancs avec des objets pandas
Vous pouvez également remplir les espaces avec un dictionnaire ou une série alignable. La clé de dictionnaire ou l'index de série doit correspondre au nom de colonne du cadre que vous souhaitez remplir. L'exemple suivant remplit le bloc de données avec la moyenne de cette colonne.
In [**]: dff = pd.DataFrame(np.random.randn(10, 3), columns=list('ABC'))
In [**]: dff.iloc[3:5, 0] = np.nan
In [**]: dff.iloc[4:6, 1] = np.nan
In [**]: dff.iloc[5:8, 2] = np.nan
In [**]: dff
Out[**]:
A B C
0 0.271860 -0.424972 0.567020
1 0.276232 -1.087401 -0.673690
2 0.113648 -1.478427 0.524988
3 NaN 0.577046 -1.715002
4 NaN NaN -1.157892
5 -1.344312 NaN NaN
6 -0.109050 1.643563 NaN
7 0.357021 -0.674600 NaN
8 -0.968914 -1.294524 0.413738
9 0.276662 -0.472035 -0.013960
In [**]: dff.fillna(dff.mean())
Out[**]:
A B C
0 0.271860 -0.424972 0.567020
1 0.276232 -1.087401 -0.673690
2 0.113648 -1.478427 0.524988
3 -0.140857 0.577046 -1.715002
4 -0.140857 -0.401419 -1.157892
5 -1.344312 -0.401419 -0.293543
6 -0.109050 1.643563 -0.293543
7 0.357021 -0.674600 -0.293543
8 -0.968914 -1.294524 0.413738
9 0.276662 -0.472035 -0.013960
In [**]: dff.fillna(dff.mean()['B':'C'])
Out[**]:
A B C
0 0.271860 -0.424972 0.567020
1 0.276232 -1.087401 -0.673690
2 0.113648 -1.478427 0.524988
3 NaN 0.577046 -1.715002
4 NaN -0.401419 -1.157892
5 -1.344312 -0.401419 -0.293543
6 -0.109050 1.643563 -0.293543
7 0.357021 -0.674600 -0.293543
8 -0.968914 -1.294524 0.413738
9 0.276662 -0.472035 -0.013960
Le résultat est le même que ci-dessus, mais dans les cas suivants, les valeurs de la série «Remplir» sont alignées.
In [**]: dff.where(pd.notna(dff), dff.mean(), axis='columns')
Out[**]:
A B C
0 0.271860 -0.424972 0.567020
1 0.276232 -1.087401 -0.673690
2 0.113648 -1.478427 0.524988
3 -0.140857 0.577046 -1.715002
4 -0.140857 -0.401419 -1.157892
5 -1.344312 -0.401419 -0.293543
6 -0.109050 1.643563 -0.293543
7 0.357021 -0.674600 -0.293543
8 -0.968914 -1.294524 0.413738
9 0.276662 -0.472035 -0.013960
Suppression des étiquettes d'axe avec des données manquantes
Vous voudrez peut-être simplement exclure de l'ensemble de données les étiquettes qui font référence aux données manquantes. Pour ce faire, dropna () < Utilisez / code>.
In [**]: df
Out[**]:
one two three
a NaN -0.282863 -1.509059
c NaN 1.212112 -0.173215
e NaN 0.000000 0.000000
f NaN 0.000000 0.000000
h NaN -0.706771 -1.039575
In [**]: df.dropna(axis=0)
Out[**]:
Empty DataFrame
Columns: [one, two, three]
Index: []
In [**]: df.dropna(axis=1)
Out[**]:
two three
a -0.282863 -1.509059
c 1.212112 -0.173215
e 0.000000 0.000000
f 0.000000 0.000000
h -0.706771 -1.039575
In [**]: df['one'].dropna()
Out[**]: Series([], Name: one, dtype: float64)
dropna () </ code> est fourni. DataFrame.dropna a considérablement plus d'options que Series.dropna, sous API (https://pandas.pydata.org/pandas-docs/stable/reference/frame.html#api-dataframe-missing) Vous pouvez le rechercher.
interpolation
A partir de la _ version 0.23.0 _: l'argument mot-clé limit_area a été ajouté.
[interpolate ()](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.DataFrame.interpolate.html#pandas.DataFrame. Pour les objets série et dataframe. Il y a interpolation) </ code>, et par défaut il effectue une interpolation linéaire sur les points de données manquants.
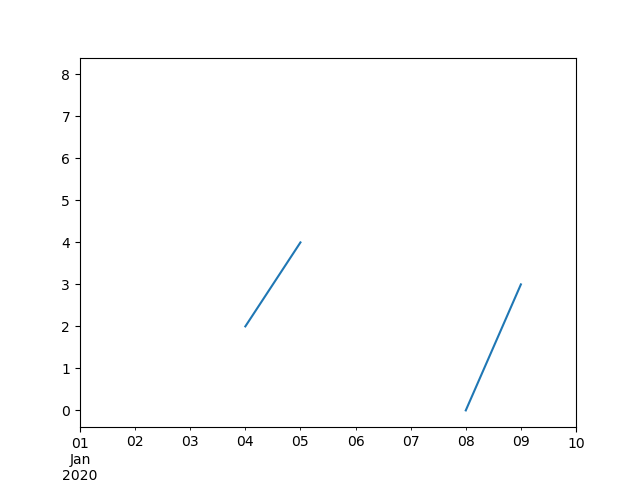
In [**]: ts
Out[**]:
2000-01-31 0.469112
2000-02-29 NaN
2000-03-31 NaN
2000-04-28 NaN
2000-05-31 NaN
...
2007-12-31 -6.950267
2008-01-31 -7.904475
2008-02-29 -6.441779
2008-03-31 -8.184940
2008-04-30 -9.011531
Freq: BM, Length: 100, dtype: float64
In [**]: ts.count()
Out[**]: 66
In [**]: ts.plot()
Out[**]: <matplotlib.axes._subplots.AxesSubplot at 0x7fc18e5ac400>
In [**]: ts.interpolate()
Out[**]:
2000-01-31 0.469112
2000-02-29 0.434469
2000-03-31 0.399826
2000-04-28 0.365184
2000-05-31 0.330541
...
2007-12-31 -6.950267
2008-01-31 -7.904475
2008-02-29 -6.441779
2008-03-31 -8.184940
2008-04-30 -9.011531
Freq: BM, Length: 100, dtype: float64
In [**]: ts.interpolate().count()
Out[**]: 100
In [**]: ts.interpolate().plot()
Out[**]: <matplotlib.axes._subplots.AxesSubplot at 0x7fc18e569880>

Vous pouvez effectuer une interpolation basée sur un index en utilisant le mot-clé method.
In [**]: ts2
Out[**]:
2000-01-31 0.469112
2000-02-29 NaN
2002-07-31 -5.785037
2005-01-31 NaN
2008-04-30 -9.011531
dtype: float64
In [**]: ts2.interpolate()
Out[**]:
2000-01-31 0.469112
2000-02-29 -2.657962
2002-07-31 -5.785037
2005-01-31 -7.398284
2008-04-30 -9.011531
dtype: float64
In [**]: ts2.interpolate(method='time')
Out[**]:
2000-01-31 0.469112
2000-02-29 0.270241
2002-07-31 -5.785037
2005-01-31 -7.190866
2008-04-30 -9.011531
dtype: float64
Pour les index à virgule flottante, utilisez method = 'values'.
In [**]: ser
Out[**]:
0.0 0.0
1.0 NaN
10.0 10.0
dtype: float64
In [**]: ser.interpolate()
Out[**]:
0.0 0.0
1.0 5.0
10.0 10.0
dtype: float64
In [**]: ser.interpolate(method='values')
Out[**]:
0.0 0.0
1.0 1.0
10.0 10.0
dtype: float64
De même, vous pouvez interpoler des blocs de données.
In [**]: df = pd.DataFrame({'A': [1, 2.1, np.nan, 4.7, 5.6, 6.8],
....: 'B': [.25, np.nan, np.nan, 4, 12.2, 14.4]})
....:
In [**]: df
Out[**]:
A B
0 1.0 0.25
1 2.1 NaN
2 NaN NaN
3 4.7 4.00
4 5.6 12.20
5 6.8 14.40
In [**]: df.interpolate()
Out[**]:
A B
0 1.0 0.25
1 2.1 1.50
2 3.4 2.75
3 4.7 4.00
4 5.6 12.20
5 6.8 14.40
Vous pouvez effectuer une interpolation plus avancée en utilisant l'argument method. Si vous avez installé scipy, vous pouvez passer le nom de la routine d'interpolation unidimensionnelle à method. Pour plus d'informations, voir Documentation et Reference [Guide](https: //docs.scipy. Voir org / doc / scipy / reference / tutorial / interpolate.html). La méthode d'interpolation appropriée dépend du type de données avec lesquelles vous travaillez.
- Lorsqu'il s'agit de séries chronologiques avec des taux de croissance élevés, «method =« quadratic »peut être approprié.
--Pour les valeurs proches de la fonction de distribution cumulative,
method = 'pchip'devrait fonctionner. - Considérez
method = 'akima'pour remplir les valeurs manquantes pour un tracé fluide.
: avertissement: ** Avertissement ** Ces méthodes nécessitent «scipy».
In [**]: df.interpolate(method='barycentric')
Out[**]:
A B
0 1.00 0.250
1 2.10 -7.660
2 3.53 -4.515
3 4.70 4.000
4 5.60 12.200
5 6.80 14.400
In [**]: df.interpolate(method='pchip')
Out[**]:
A B
0 1.00000 0.250000
1 2.10000 0.672808
2 3.43454 1.928950
3 4.70000 4.000000
4 5.60000 12.200000
5 6.80000 14.400000
In [**]: df.interpolate(method='akima')
Out[**]:
A B
0 1.000000 0.250000
1 2.100000 -0.873316
2 3.406667 0.320034
3 4.700000 4.000000
4 5.600000 12.200000
5 6.800000 14.400000
Lors de l'interpolation avec une approximation polymorphe ou spline, vous devez également spécifier l'ordre de l'approximation.
In [**]: df.interpolate(method='spline', order=2)
Out[**]:
A B
0 1.000000 0.250000
1 2.100000 -0.428598
2 3.404545 1.206900
3 4.700000 4.000000
4 5.600000 12.200000
5 6.800000 14.400000
In [**]: df.interpolate(method='polynomial', order=2)
Out[**]:
A B
0 1.000000 0.250000
1 2.100000 -2.703846
2 3.451351 -1.453846
3 4.700000 4.000000
4 5.600000 12.200000
5 6.800000 14.400000
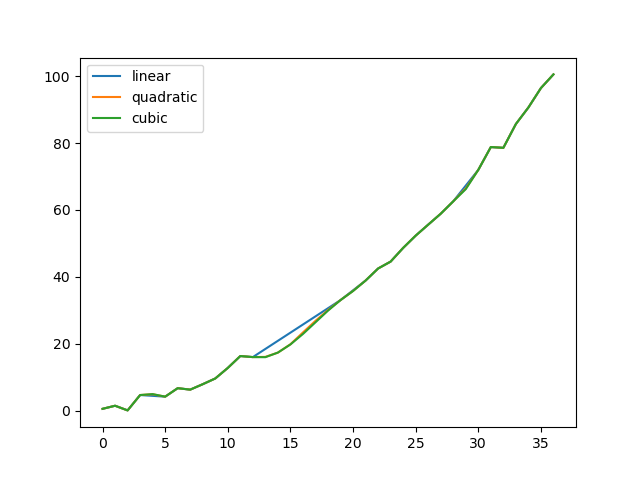
Comparons plusieurs méthodes.
In [**]: np.random.seed(2)
In [**]: ser = pd.Series(np.arange(1, 10.1, .25) ** 2 + np.random.randn(37))
In [**]: missing = np.array([4, 13, 14, 15, 16, 17, 18, 20, 29])
In [**]: ser[missing] = np.nan
In [**]: methods = ['linear', 'quadratic', 'cubic']
In [**]: df = pd.DataFrame({m: ser.interpolate(method=m) for m in methods})
In [**]: df.plot()
Out[**]: <matplotlib.axes._subplots.AxesSubplot at 0x7fc18e5b6c70>
Un autre cas d'utilisation est l'interpolation avec de nouvelles valeurs. Supposons que vous ayez 100 observations à partir d'une distribution. Et disons que vous êtes particulièrement intéressé par ce qui se passe près du centre. Vous pouvez interpoler avec de nouvelles valeurs en combinant les méthodes reindex et ʻinterpolate` des pandas.
In [**]: ser = pd.Series(np.sort(np.random.uniform(size=100)))
#Interpolation pour un nouvel index
In [**]: new_index = ser.index | pd.Index([49.25, 49.5, 49.75, 50.25, 50.5, 50.75])
In [**]: interp_s = ser.reindex(new_index).interpolate(method='pchip')
In [**]: interp_s[49:51]
Out[**]:
49.00 0.471410
49.25 0.476841
49.50 0.481780
49.75 0.485998
50.00 0.489266
50.25 0.491814
50.50 0.493995
50.75 0.495763
51.00 0.497074
dtype: float64
Limites d'interpolation
Comme les autres méthodes de remplissage des blancs pandas, [interpolate ()](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.DataFrame.interpolate.html#pandas.DataFrame .interpolate) </ code> prend l'argument de mot-clé limit. Vous pouvez utiliser cet argument pour limiter le nombre de valeurs «NaN» consécutives qui ont été entrées depuis la dernière observation valide.
In [**]: ser = pd.Series([np.nan, np.nan, 5, np.nan, np.nan,
....: np.nan, 13, np.nan, np.nan])
....:
In [**]: ser
Out[**]:
0 NaN
1 NaN
2 5.0
3 NaN
4 NaN
5 NaN
6 13.0
7 NaN
8 NaN
dtype: float64
#Remplissez toutes les valeurs d'affilée vers l'avant
In [**]: ser.interpolate()
Out[**]:
0 NaN
1 NaN
2 5.0
3 7.0
4 9.0
5 11.0
6 13.0
7 13.0
8 13.0
dtype: float64
#Remplissez une seule valeur avant
In [**]: ser.interpolate(limit=1)
Out[**]:
0 NaN
1 NaN
2 5.0
3 7.0
4 NaN
5 NaN
6 13.0
7 13.0
8 NaN
dtype: float64
Par défaut, la valeur NaN est remplie en avant. Utilisez le paramètre limit_direction pour remplir les trous par l'arrière ou dans les deux sens.
#Remplissez un trou à l'envers
In [**]: ser.interpolate(limit=1, limit_direction='backward')
Out[**]:
0 NaN
1 5.0
2 5.0
3 NaN
4 NaN
5 11.0
6 13.0
7 NaN
8 NaN
dtype: float64
#Remplissez un trou dans les deux sens
In [**]: ser.interpolate(limit=1, limit_direction='both')
Out[**]:
0 NaN
1 5.0
2 5.0
3 7.0
4 NaN
5 11.0
6 13.0
7 13.0
8 NaN
dtype: float64
#Remplissez toutes les valeurs consécutives dans les deux sens
In [**]: ser.interpolate(limit_direction='both')
Out[**]:
0 5.0
1 5.0
2 5.0
3 7.0
4 9.0
5 11.0
6 13.0
7 13.0
8 13.0
dtype: float64
Par défaut, la valeur NaN est remplie à l'intérieur (entre les parties) de la valeur valide existante ou à l'extérieur de la valeur valide existante. Le paramètre limit_area introduit dans la v0.23 limite l'entrée à des valeurs internes ou externes.
#Remplissez une valeur intérieure continue dans les deux sens
In [**]: ser.interpolate(limit_direction='both', limit_area='inside', limit=1)
Out[**]:
0 NaN
1 NaN
2 5.0
3 7.0
4 NaN
5 11.0
6 13.0
7 NaN
8 NaN
dtype: float64
#Remplissez toutes les valeurs externes consécutives dans la direction opposée
In [**]: ser.interpolate(limit_direction='backward', limit_area='outside')
Out[**]:
0 5.0
1 5.0
2 5.0
3 NaN
4 NaN
5 NaN
6 13.0
7 NaN
8 NaN
dtype: float64
#Remplissez toutes les valeurs externes qui sont continues dans les deux directions
In [**]: ser.interpolate(limit_direction='both', limit_area='outside')
Out[**]:
0 5.0
1 5.0
2 5.0
3 NaN
4 NaN
5 NaN
6 13.0
7 13.0
8 13.0
dtype: float64
Remplacement de la valeur générale
Vous souhaitez souvent remplacer une valeur par une autre.
Série replace () </ code> et Trame de données replace () </ code> Vous permet d'effectuer de tels remplacements de manière efficace et flexible.
Pour les séries, vous pouvez remplacer une valeur unique ou une liste de valeurs par une autre valeur.
In [**]: ser = pd.Series([0., 1., 2., 3., 4.])
In [**]: ser.replace(0, 5)
Out[**]:
0 5.0
1 1.0
2 2.0
3 3.0
4 4.0
dtype: float64
Vous pouvez remplacer la liste de valeurs par une liste d'autres valeurs.
In [**]: ser.replace([0, 1, 2, 3, 4], [4, 3, 2, 1, 0])
Out[**]:
0 4.0
1 3.0
2 2.0
3 1.0
4 0.0
dtype: float64
Vous pouvez également spécifier un dictionnaire de mappage.
In [**]: ser.replace({0: 10, 1: 100})
Out[**]:
0 10.0
1 100.0
2 2.0
3 3.0
4 4.0
dtype: float64
Pour les blocs de données, vous pouvez spécifier une valeur distincte pour chaque colonne.
In [**]: df = pd.DataFrame({'a': [0, 1, 2, 3, 4], 'b': [5, 6, 7, 8, 9]})
In [**]: df.replace({'a': 0, 'b': 5}, 100)
Out[**]:
a b
0 100 100
1 1 6
2 2 7
3 3 8
4 4 9
Au lieu de remplacer par la valeur spécifiée, vous pouvez traiter toutes les valeurs spécifiées comme des valeurs manquantes et les interpoler.
In [**]: ser.replace([1, 2, 3], method='pad')
Out[**]:
0 0.0
1 0.0
2 0.0
3 0.0
4 4.0
dtype: float64
Remplacement des chaînes de caractères et des expressions régulières
: information_source: ** Remarque ** Les chaînes Python précédées de «r», telles que «r'hello world», sont des chaînes dites «brutes». Ils ont une sémantique inverse différente de celle des chaînes sans préfixe. Les barres obliques inverses dans la chaîne brute sont interprétées comme des barres obliques inverses d'échappement (par exemple,
r '\' == '\\'). Si vous n'êtes pas sûr de cela, vous devriez lire à ce sujet (https://docs.python.org/3/reference/lexical_analysis.html#string-literals).
Remplacez «.» Par «NaN» (chaîne de caractères → chaîne de caractères).
In [**]: d = {'a': list(range(4)), 'b': list('ab..'), 'c': ['a', 'b', np.nan, 'd']}
In [**]: df = pd.DataFrame(d)
In [**]: df.replace('.', np.nan)
Out[**]:
a b c
0 0 a a
1 1 b b
2 2 NaN NaN
3 3 NaN d
Ensuite, supprimez les espaces environnants avec l'expression régulière (expression régulière → expression régulière).
In [**]: df.replace(r'\s*\.\s*', np.nan, regex=True)
Out[**]:
a b c
0 0 a a
1 1 b b
2 2 NaN NaN
3 3 NaN d
Remplacez certaines valeurs différentes (liste → liste).
In [**]: df.replace(['a', '.'], ['b', np.nan])
Out[**]:
a b c
0 0 b b
1 1 b b
2 2 NaN NaN
3 3 NaN d
Liste des expressions régulières → Liste des expressions régulières.
In [**]: df.replace([r'\.', r'(a)'], ['dot', r'\1stuff'], regex=True)
Out[**]:
a b c
0 0 astuff astuff
1 1 b b
2 2 dot NaN
3 3 dot d
Rechercher uniquement la colonne `` b '' (Dictionnaire → Dictionnaire).
In [**]: df.replace({'b': '.'}, {'b': np.nan})
Out[**]:
a b c
0 0 a a
1 1 b b
2 2 NaN NaN
3 3 NaN d
Identique à l'exemple précédent, mais utilisez plutôt une expression régulière pour la recherche (dictionnaire d'expressions régulières → dictionnaire).
In [**]: df.replace({'b': r'\s*\.\s*'}, {'b': np.nan}, regex=True)
Out[**]:
a b c
0 0 a a
1 1 b b
2 2 NaN NaN
3 3 NaN d
Vous pouvez utiliser regex = True pour transmettre un dictionnaire imbriqué d'expressions régulières.
In [**]: df.replace({'b': {'b': r''}}, regex=True)
Out[**]:
a b c
0 0 a a
1 1 b
2 2 . NaN
3 3 . d
Vous pouvez également transmettre un dictionnaire imbriqué comme suit:
In [**]: df.replace(regex={'b': {r'\s*\.\s*': np.nan}})
Out[**]:
a b c
0 0 a a
1 1 b b
2 2 NaN NaN
3 3 NaN d
Vous pouvez également remplacer à l'aide d'un groupe de correspondances d'expressions régulières (Dictionnaire d'expressions régulières → Dictionnaire d'expressions régulières). Cela fonctionne également pour les listes.
In [**]: df.replace({'b': r'\s*(\.)\s*'}, {'b': r'\1ty'}, regex=True)
Out[**]:
a b c
0 0 a a
1 1 b b
2 2 .ty NaN
3 3 .ty d
Vous pouvez passer une liste d'expressions régulières, et toutes les correspondances seront remplacées par des scalaires (liste d'expressions régulières → expressions régulières).
In [**]: df.replace([r'\s*\.\s*', r'a|b'], np.nan, regex=True)
Out[**]:
a b c
0 0 NaN NaN
1 1 NaN NaN
2 2 NaN NaN
3 3 NaN d
Tous les exemples d'expressions régulières peuvent également passer l'argument to_replace comme argument regex. Dans ce cas, l'argument «value» doit être explicitement passé par nom, ou «regex» doit être un dictionnaire imbriqué. L'exemple précédent dans ce cas ressemble à ceci:
In [**]: df.replace(regex=[r'\s*\.\s*', r'a|b'], value=np.nan)
Out[**]:
a b c
0 0 NaN NaN
1 1 NaN NaN
2 2 NaN NaN
3 3 NaN d
Ceci est utile si vous ne voulez pas passer regex = True à chaque fois que vous utilisez une expression régulière.
Dans l'exemple
replaceci-dessus, partout où vous pouvez passer une expression régulière, transmettre une expression régulière compilée est tout aussi valide.
Remplacement numérique
replace () </ code> est > Similaire à fillna () </ code>.
In [**]: df = pd.DataFrame(np.random.randn(10, 2))
In [**]: df[np.random.rand(df.shape[0]) > 0.5] = 1.5
In [**]: df.replace(1.5, np.nan)
Out[**]:
0 1
0 -0.844214 -1.021415
1 0.432396 -0.323580
2 0.423825 0.799180
3 1.262614 0.751965
4 NaN NaN
5 NaN NaN
6 -0.498174 -1.060799
7 0.591667 -0.183257
8 1.019855 -1.482465
9 NaN NaN
Vous pouvez remplacer plusieurs valeurs en passant une liste.
In [**]: df00 = df.iloc[0, 0]
In [**]: df.replace([1.5, df00], [np.nan, 'a'])
Out[**]:
0 1
0 a -1.02141
1 0.432396 -0.32358
2 0.423825 0.79918
3 1.26261 0.751965
4 NaN NaN
5 NaN NaN
6 -0.498174 -1.0608
7 0.591667 -0.183257
8 1.01985 -1.48247
9 NaN NaN
In [**]: df[1].dtype
Out[**]: dtype('float64')
Vous pouvez également traiter la trame de données sur place.
In [**]: df.replace(1.5, np.nan, inplace=True)
: avertissement: ** Avertissement ** Lors du remplacement de plusieurs objets
booloudatetime64, le premier argument dereplace(to_replace) doit correspondre au type de la valeur à remplacer. Par exemple>>> s = pd.Series([True, False, True]) >>> s.replace({'a string': 'new value', True: False}) # raises TypeError: Cannot compare types 'ndarray(dtype=bool)' and 'str'Une
TypeErrorse produit car l'une des clésdictn'est pas un type approprié pour le remplacement.Cependant, si vous souhaitez remplacer un * seul * objet comme celui-ci:
In [**]: s = pd.Series([True, False, True]) In [**]: s.replace('a string', 'another string') Out[**]: 0 True 1 False 2 True dtype: boolL'objet
NDFramed'origine est renvoyé tel quel. Nous travaillons actuellement sur l'intégration de cette API, mais pour des raisons de compatibilité descendante, nous ne pouvons pas casser ce dernier comportement. Pour plus d'informations, consultez GH6354.
Règles de conversion et indexation des données manquantes
pandas prend en charge le stockage de tableaux entiers et booléens, mais ces types ne peuvent pas stocker les données manquantes. Jusqu'à ce que NumPy passe à l'utilisation du type NA natif, nous avions mis en place des «règles de distribution». Si l'opération de réindexation entraîne des données manquantes, la série sera castée selon les règles présentées dans le tableau ci-dessous.
| Type de données | Destination de diffusion |
|---|---|
| entier | Fraction flottante |
| Valeur booléenne | objet |
| Fraction flottante | Ne lancez pas |
| objet | Ne lancez pas |
Par exemple
In [**]: s = pd.Series(np.random.randn(5), index=[0, 2, 4, 6, 7])
In [**]: s > 0
Out[**]:
0 True
2 True
4 True
6 True
7 True
dtype: bool
In [**]: (s > 0).dtype
Out[**]: dtype('bool')
In [**]: crit = (s > 0).reindex(list(range(8)))
In [**]: crit
Out[**]:
0 True
1 NaN
2 True
3 NaN
4 True
5 NaN
6 True
7 True
dtype: object
In [**]: crit.dtype
Out[**]: dtype('O')
Normalement, NumPy se plaint en essayant d'obtenir ou de définir une valeur à partir d'un ndarray en utilisant un tableau d'objets au lieu d'un tableau booléen (par exemple, en choisissant une valeur basée sur certains critères). Une exception est déclenchée si le vecteur booléen contient NA.
In [**]: reindexed = s.reindex(list(range(8))).fillna(0)
In [**]: reindexed[crit]
---------------------------------------------------------------------------
ValueError Traceback (most recent call last)
<ipython-input-138-0dac417a4890> in <module>
----> 1 reindexed[crit]
~/work/pandas/pandas/pandas/core/series.py in __getitem__(self, key)
901 key = list(key)
902
--> 903 if com.is_bool_indexer(key):
904 key = check_bool_indexer(self.index, key)
905 key = np.asarray(key, dtype=bool)
~/work/pandas/pandas/pandas/core/common.py in is_bool_indexer(key)
132 na_msg = "Cannot mask with non-boolean array containing NA / NaN values"
133 if isna(key).any():
--> 134 raise ValueError(na_msg)
135 return False
136 return True
ValueError: Cannot mask with non-boolean array containing NA / NaN values
Cependant, il s'agit de fillna () </ code Si vous utilisez> pour remplir les trous, cela fonctionne très bien.
In [**]: reindexed[crit.fillna(False)]
Out[**]:
0 0.126504
2 0.696198
4 0.697416
6 0.601516
7 0.003659
dtype: float64
In [**]: reindexed[crit.fillna(True)]
Out[**]:
0 0.126504
1 0.000000
2 0.696198
3 0.000000
4 0.697416
5 0.000000
6 0.601516
7 0.003659
dtype: float64
pandas fournit des types de données entiers qui peuvent avoir des valeurs manquantes, mais vous devez les spécifier explicitement lors de la création d'une série ou d'une colonne. Notez que nous utilisons le "I" supérieur dans dtype =" Int64 ".
In [**]: s = pd.Series([0, 1, np.nan, 3, 4], dtype="Int64")
In [**]: s
Out[**]:
0 0
1 1
2 <NA>
3 3
4 4
dtype: Int64
Voir Types de données entiers manquants (https://pandas.pydata.org/pandas-docs/stable/user_guide/integer_na.html#integer-na) pour plus d'informations.
Scalaire expérimental «NA» pour afficher les valeurs manquantes
: avertissement: ** Avertissement ** Test de fonctionnement: Le comportement de
pd.NAest sujet à changement sans avertissement.
_ À partir de la version 1.0.0 _
À partir de pandas 1.0, la valeur expérimentale pd.NA (une tonne) est disponible pour représenter les valeurs scalaires manquantes. Actuellement, les types de données Entier Nullable, Booléen et Chaîne dédiée sont utilisés comme indicateurs de valeur manquante.
À partir de pandas 1.0, les valeurs pd.NA expérimentales (tonnes uniques) peuvent être utilisées pour représenter les valeurs scalaires manquantes. Pour le moment, Missable Integer, Bourian et [Dedicated String](https: // qiita.com/nkay/items/3866b3f12704ec3271ca # Type de données texte) Il est utilisé comme indicateur de valeur manquante dans le type de données.
Le but de «pd.NA» est de fournir des valeurs «manquantes» qui peuvent être utilisées de manière cohérente entre les types de données (au lieu de «np.nan», «None», «pd.NaT» selon le type de données À).
Par exemple, s'il y a des valeurs manquantes dans une série de types de données entiers manquants, pd.NA est utilisé.
In [**]: s = pd.Series([1, 2, None], dtype="Int64")
In [**]: s
Out[**]:
0 1
1 2
2 <NA>
dtype: Int64
In [**]: s[2]
Out[**]: <NA>
In [**]: s[2] is pd.NA
Out[**]: True
Actuellement, pandas n'utilise pas encore ces types de données par défaut (lors de la création ou du chargement de trames de données ou de séries), vous devez donc spécifier explicitement les types de données. Un moyen simple de convertir vers ces types de données est décrit ici (# conversion).
Propagation dans les opérations arithmétiques et de comparaison
En général, les opérations impliquant «pd.NA» propagent les valeurs manquantes. Si l'un des opérandes est inconnu, le résultat de l'opération est également inconnu.
Par exemple, «pd.NA» se propage arithmétiquement comme «np.nan».
In [**]: pd.NA + 1
Out[**]: <NA>
In [**]: "a" * pd.NA
Out[**]: <NA>
Même si l'un des opérandes est "NA", il existe des cas particuliers si le résultat est connu.
In [**]: pd.NA ** 0
Out[**]: 1
In [**]: 1 ** pd.NA
Out[**]: 1
Les opérations d'égalité et de comparaison propagent également pd.NA. Ceci est différent du comportement de «np.nan», où le résultat de la comparaison est toujours «False».
In [**]: pd.NA == 1
Out[**]: <NA>
In [**]: pd.NA == pd.NA
Out[**]: <NA>
In [**]: pd.NA < 2.5
Out[**]: <NA>
Pour savoir si la valeur est égale à pd.NA, [isna ()](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.isna.html Utilisez la fonction # pandas.isna) </ code>.
In [**]: pd.isna(pd.NA)
Out[**]: True
L'exception à cette règle de propagation de base est la * réduction * (comme la moyenne ou le minimum), et les pandas ignorent les valeurs manquantes par défaut. Pour plus de détails, consultez [ci-dessus](## Calcul des données manquantes).
Opération logique
Pour les opérations logiques, pd.NA est [logique ternaire](https://ja.wikipedia.org/wiki/3value logic) (également appelée logique de Kleine, qui se comporte comme R / SQL / Julia) Suivez les règles de. Cette logique signifie la propagation des valeurs manquantes uniquement lorsque cela est logiquement nécessaire.
Par exemple, dans le cas de l'opération logique "ou" ("|"), si l'un des opérandes est "True", il est "False" quelle que soit l'autre valeur (c'est-à-dire même si la valeur manquante est "True" ". Aussi), nous savons déjà que le résultat sera "True". Dans ce cas, pd.NA ne se propagera pas.
In [**]: True | False
Out[**]: True
In [**]: True | pd.NA
Out[**]: True
In [**]: pd.NA | True
Out[**]: True
D'un autre côté, si l'un des opérandes est "False", le résultat dépend de la valeur de l'autre opérande. Par conséquent, dans ce cas, pd.NA se propage.
In [**]: False | True
Out[**]: True
In [**]: False | False
Out[**]: False
In [**]: False | pd.NA
Out[**]: <NA>
Le comportement de l'opération logique «et» («&») peut également être dérivé en utilisant une logique similaire (dans ce cas, «pd.NA» si l'un des opérandes est déjà «Faux». Ne se propage pas).
In [**]: False & True
Out[**]: False
In [**]: False & False
Out[**]: False
In [**]: False & pd.NA
Out[**]: False
In [**]: True & True
Out[**]: True
In [**]: True & False
Out[**]: False
In [**]: True & pd.NA
Out[**]: <NA>
NA dans le contexte bourian
Les valeurs manquantes ne peuvent pas être converties en valeurs booléennes car la valeur réelle est inconnue. Ce qui suit entraînera une erreur.
In [**]: bool(pd.NA)
---------------------------------------------------------------------------
TypeError Traceback (most recent call last)
<ipython-input-167-5477a57d5abb> in <module>
----> 1 bool(pd.NA)
~/work/pandas/pandas/pandas/_libs/missing.pyx in pandas._libs.missing.NAType.__bool__()
TypeError: boolean value of NA is ambiguous
Cela signifie également que dans un contexte où «pd.NA» est évalué avec une valeur booléenne, par exemple «si condition: ...», où «condition» peut être «pd.NA», alors «pd. NA »signifie qu'il ne peut pas être utilisé. Dans ce cas, utilisez isna () </ code> Vous pouvez l'utiliser pour vérifier «pd.NA» ou, par exemple, préremplir les valeurs manquantes pour éviter que la condition ne soit «pd.NA».
Une situation similaire se produit lors de l'utilisation d'objets série ou dataframe dans l'instruction ʻif`. Voir Utiliser des déclarations if / Truth avec des pandas (https://pandas.pydata.org/pandas-docs/stable/user_guide/gotchas.html#gotchas-truth).
NumPy ufunc
pandas.NA implémente le protocole array_ufunc` de NumPy. La plupart des ufunc fonctionnent pour «NA» et retournent généralement «NA».
In [**]: np.log(pd.NA)
Out[**]: <NA>
In [**]: np.add(pd.NA, 1)
Out[**]: <NA>
: avertissement: ** Avertissement ** Actuellement, ufunc avec ndarray et
NArenvoie un type de données d'objet rempli de valeurs manquantes.In [**]: a = np.array([1, 2, 3]) In [**]: np.greater(a, pd.NA) Out[**]: array([<NA>, <NA>, <NA>], dtype=object)Le type de retour ici peut être modifié pour renvoyer un autre type de tableau à l'avenir.
Pour plus d'informations sur ufunc, consultez Interopérabilité des fonctions DataFrame et NumPy (https://pandas.pydata.org/pandas-docs/stable/user_guide/dsintro.html#dsintro-numpy-interop) ..
conversion
Si vous avez un bloc de données ou une série qui utilise des types traditionnels et que vous avez des données qui ne sont pas représentées avec np.nan, la série aura [convert_dtypes ()](https: //). pandas.pydata.org/pandas-docs/stable/reference/api/pandas.Series.convert_dtypes.html#pandas.Series.convert_dtypes) </ code>, mais convert_dtypes () dans le cadre de données Il y a //pandas.pydata.org/pandas-docs/stable/reference/api/pandas.DataFrame.convert_dtypes.html#pandas.DataFrame.convert_dtypes) </ code> et [ici](https: // pandas. Vous pouvez convertir vos données pour utiliser les nouveaux types de données d'entiers, de chaînes et de bourians répertoriés dans pydata.org/pandas-docs/stable/user_guide/basics.html#basics-dtypes). Il s'agit de read_csv () après le chargement de l'ensemble de données. Pour les lecteurs tels que code> et read_excel () </ code> Ceci est particulièrement utile pour deviner le type de données par défaut.
Dans cet exemple, les types de données de toutes les colonnes ont changé, mais les résultats des 10 premières colonnes sont affichés.
In [**]: bb = pd.read_csv('data/baseball.csv', index_col='id')
In [**]: bb[bb.columns[:10]].dtypes
Out[**]:
player object
year int64
stint int64
team object
lg object
g int64
ab int64
r int64
h int64
X2b int64
dtype: object
In [**]: bbn = bb.convert_dtypes()
In [**]: bbn[bbn.columns[:10]].dtypes
Out[**]:
player string
year Int64
stint Int64
team string
lg string
g Int64
ab Int64
r Int64
h Int64
X2b Int64
dtype: object
Recommended Posts