Guide de l'utilisateur de Pandas "Formatage de tableau et tableau croisé dynamique" (Document officiel traduction japonaise)
Cet article est une traduction automatique partielle de la documentation officielle de Pandas Guide de l'utilisateur - Remodelage et tableaux croisés dynamiques. C'est une modification d'une phrase contre nature.
Si vous avez des erreurs de traduction, des traductions alternatives, des questions, etc., veuillez utiliser la section commentaires ou modifier la demande.
Mise en forme du tableau et tableau croisé dynamique
Mise en forme en faisant pivoter un objet DataFrame
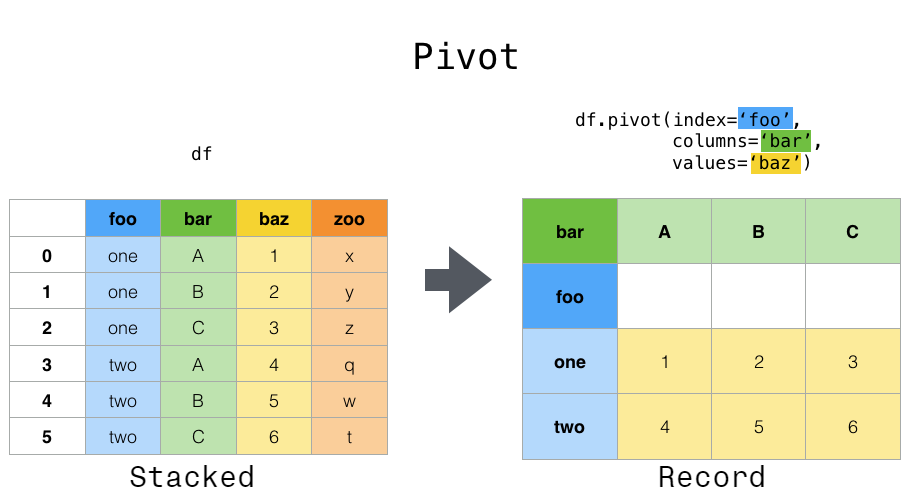
Les données sont souvent stockées dans ce que l'on appelle des «piles» ou des «enregistrements».
In [1]: df
Out[1]:
date variable value
0 2000-01-03 A 0.469112
1 2000-01-04 A -0.282863
2 2000-01-05 A -1.509059
3 2000-01-03 B -1.135632
4 2000-01-04 B 1.212112
5 2000-01-05 B -0.173215
6 2000-01-03 C 0.119209
7 2000-01-04 C -1.044236
8 2000-01-05 C -0.861849
9 2000-01-03 D -2.104569
10 2000-01-04 D -0.494929
11 2000-01-05 D 1.071804
À propos, comment créer le DataFrame ci-dessus est la suivante.
import pandas._testing as tm
def unpivot(frame):
N, K = frame.shape
data = {'value': frame.to_numpy().ravel('F'),
'variable': np.asarray(frame.columns).repeat(N),
'date': np.tile(np.asarray(frame.index), K)}
return pd.DataFrame(data, columns=['date', 'variable', 'value'])
df = unpivot(tm.makeTimeDataFrame(3))
Pour sélectionner toutes les lignes où la variable est ʻA`:
In [2]: df[df['variable'] == 'A']
Out[2]:
date variable value
0 2000-01-03 A 0.469112
1 2000-01-04 A -0.282863
2 2000-01-05 A -1.509059
En revanche, supposons que vous souhaitiez utiliser ces variables pour effectuer des opérations de séries chronologiques. Dans ce cas, il serait préférable d'identifier les observations individuelles par la variable unique «colonnes» et la date «index». Pour remodeler les données dans ce format, [DataFrame.pivot ()](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.DataFrame.pivot.html#pandas Utilisez la méthode .DataFrame.pivot) (fonction de niveau supérieur pivot () .pivot) est également implémenté).
In [3]: df.pivot(index='date', columns='variable', values='value')
Out[3]:
variable A B C D
date
2000-01-03 0.469112 -1.135632 0.119209 -2.104569
2000-01-04 -0.282863 1.212112 -1.044236 -0.494929
2000-01-05 -1.509059 -0.173215 -0.861849 1.071804
Si l'argument values est omis et que l'entrée DataFrame a plusieurs colonnes de valeurs qui ne sont pas données à pivot comme colonnes ou index, le "pivot" résultant DataFrame aura le niveau le plus élevé de chaque colonne de valeurs. A une colonne hiérarchique (https://qiita.com/nkay/items/ Multi-Index Advanced Index) qui indique.
In [4]: df['value2'] = df['value'] * 2
In [5]: pivoted = df.pivot(index='date', columns='variable')
In [6]: pivoted
Out[6]:
value ... value2
variable A B C ... B C D
date ...
2000-01-03 0.469112 -1.135632 0.119209 ... -2.271265 0.238417 -4.209138
2000-01-04 -0.282863 1.212112 -1.044236 ... 2.424224 -2.088472 -0.989859
2000-01-05 -1.509059 -0.173215 -0.861849 ... -0.346429 -1.723698 2.143608
[3 rows x 8 columns]
Vous pouvez ensuite sélectionner un sous-ensemble dans le DataFrame pivoté.
In [7]: pivoted['value2']
Out[7]:
variable A B C D
date
2000-01-03 0.938225 -2.271265 0.238417 -4.209138
2000-01-04 -0.565727 2.424224 -2.088472 -0.989859
2000-01-05 -3.018117 -0.346429 -1.723698 2.143608
Notez que si les données sont du même type, elles retournent une vue des données sous-jacentes.
: ballot_box_with_check: ** Remarque ** Si la paire index / colonne n'est pas unique,
pivot ()Déclenche l'erreurValueError: Index contient des entrées en double, ne peut pas être remodelé. Dans ce cas, envisagez d'utiliserpivot_table (). S'il te plait donne moi. Il s'agit d'un pivot généralisé qui peut gérer les valeurs en double pour une seule paire index / colonne.
Changement de forme par empilement et dépilage
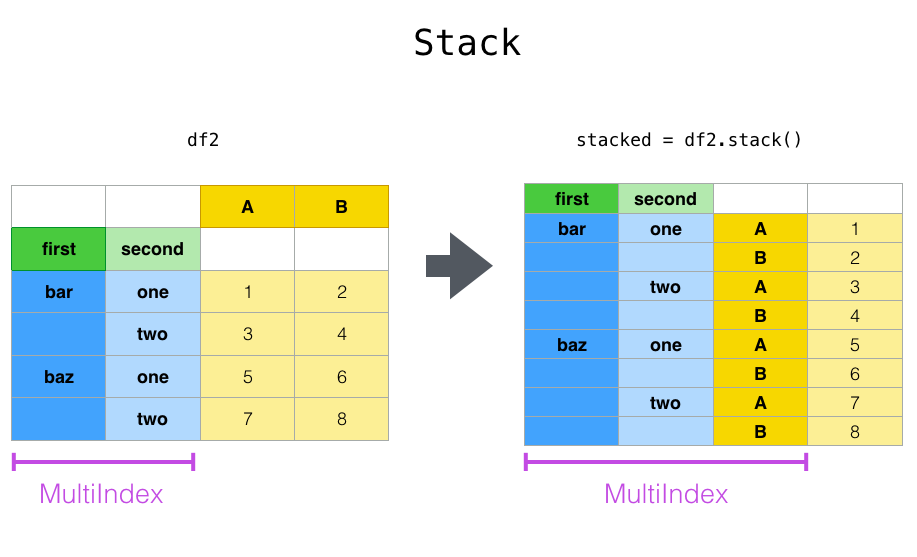
pivot () La même série étroitement liée à la méthode Stack () disponible dans Series et DataFrame à la méthode de # pandas.DataFrame.stack) et [ʻunstack () ](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.DataFrame.unstack.html#pandas.DataFrame.unstack) Il existe une méthode. Ces méthodes sont conçues pour fonctionner avec l'objet MultiIndex` (voir le chapitre sur les index hiérarchiques (https://qiita.com/nkay/items/Multi-Index Advanced Indexes)). Les fonctions de base de ces méthodes sont les suivantes:
--stack: "Pivote" le niveau de l'étiquette de colonne (éventuellement hiérarchique) et renvoie un nouveau DataFrame avec cette étiquette de ligne au niveau le plus profond de l'index.
--ʻUnstack`: (empilement inversé) (probablement hiérarchique) niveau d'index de ligne "pivoter" vers l'axe de colonne pour générer un "DataFrame" reconstruit avec le libellé de colonne de niveau le plus interne Je vais.
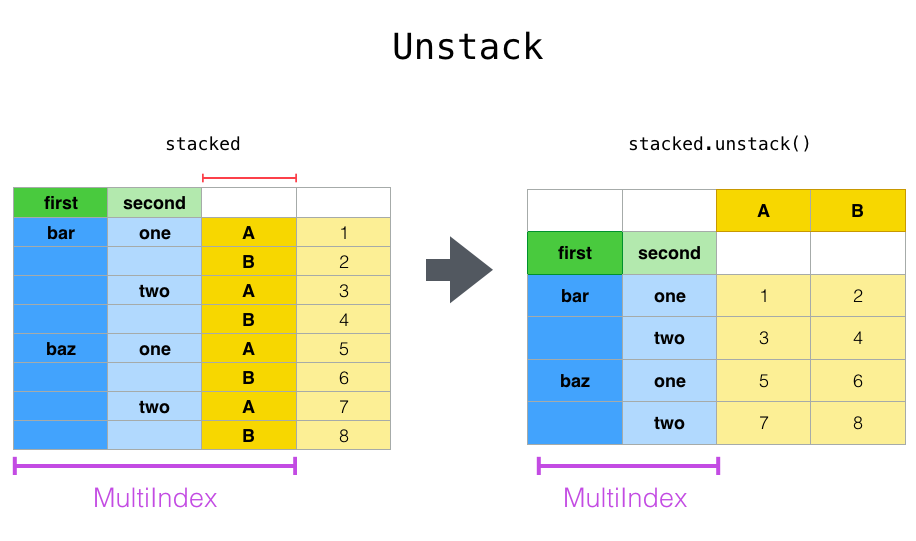
Ce sera plus facile à comprendre si vous regardez un exemple réel. Il traite du même jeu de données que celui que nous avons vu dans le chapitre Index hiérarchique.
In [8]: tuples = list(zip(*[['bar', 'bar', 'baz', 'baz',
...: 'foo', 'foo', 'qux', 'qux'],
...: ['one', 'two', 'one', 'two',
...: 'one', 'two', 'one', 'two']]))
...:
In [9]: index = pd.MultiIndex.from_tuples(tuples, names=['first', 'second'])
In [10]: df = pd.DataFrame(np.random.randn(8, 2), index=index, columns=['A', 'B'])
In [11]: df2 = df[:4]
In [12]: df2
Out[12]:
A B
first second
bar one 0.721555 -0.706771
two -1.039575 0.271860
baz one -0.424972 0.567020
two 0.276232 -1.087401
La fonction stack" compresse "le niveau de la colonne DataFrame pour produire l'un des éléments suivants:
--Series: pour les index de colonnes simples.
--DataFrame: si la colonne a MultiIndex.
Si la colonne a «MultiIndex», vous pouvez choisir le niveau à empiler. Le niveau empilé sera le niveau le plus bas de la nouvelle colonne «MultiIndex».
In [13]: stacked = df2.stack()
In [14]: stacked
Out[14]:
first second
bar one A 0.721555
B -0.706771
two A -1.039575
B 0.271860
baz one A -0.424972
B 0.567020
two A 0.276232
B -1.087401
dtype: float64
Pour un "DataFrame" ou "Series" "empilé" (autrement dit, "index" est "MultiIndex"), utilisez ʻunstack pour effectuer l'opposé de stack`. La valeur par défaut consiste à désempiler le ** niveau le plus bas **.
In [15]: stacked.unstack()
Out[15]:
A B
first second
bar one 0.721555 -0.706771
two -1.039575 0.271860
baz one -0.424972 0.567020
two 0.276232 -1.087401
In [16]: stacked.unstack(1)
Out[16]:
second one two
first
bar A 0.721555 -1.039575
B -0.706771 0.271860
baz A -0.424972 0.276232
B 0.567020 -1.087401
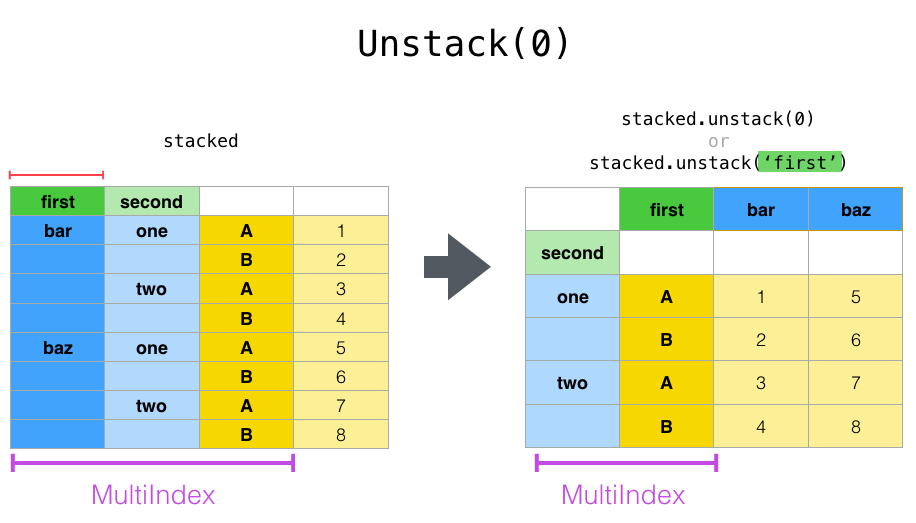
In [17]: stacked.unstack(0)
Out[17]:
first bar baz
second
one A 0.721555 -0.424972
B -0.706771 0.567020
two A -1.039575 0.276232
B 0.271860 -1.087401
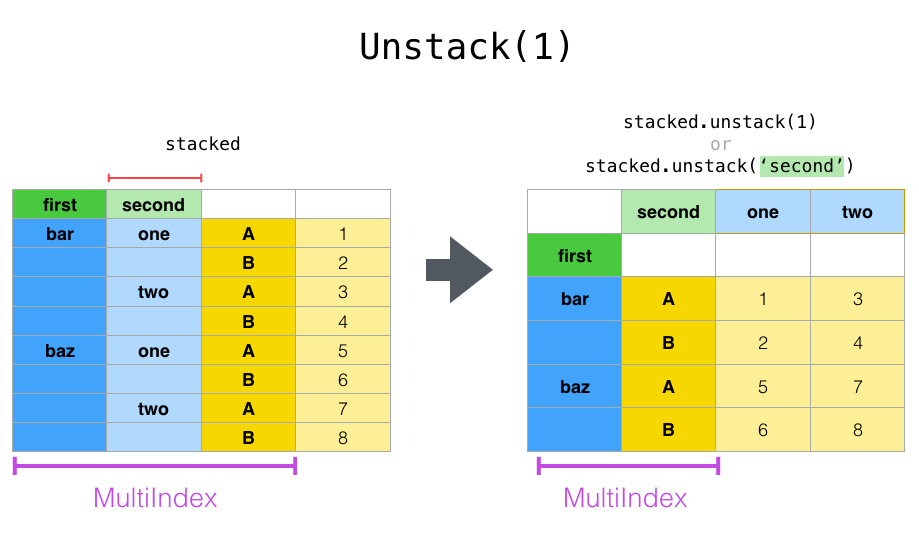
Si l'index a un nom, vous pouvez utiliser le nom du niveau au lieu de spécifier le numéro du niveau.
In [18]: stacked.unstack('second')
Out[18]:
second one two
first
bar A 0.721555 -1.039575
B -0.706771 0.271860
baz A -0.424972 0.276232
B 0.567020 -1.087401
Notez que les méthodes stack et ʻunstacktrient implicitement les niveaux d'index associés. Par conséquent,stack, puis ʻunstack (ou vice versa) renverra une copie ** triée ** du DataFrame ou de la Series d'origine.
In [19]: index = pd.MultiIndex.from_product([[2, 1], ['a', 'b']])
In [20]: df = pd.DataFrame(np.random.randn(4), index=index, columns=['A'])
In [21]: df
Out[21]:
A
2 a -0.370647
b -1.157892
1 a -1.344312
b 0.844885
In [22]: all(df.unstack().stack() == df.sort_index())
Out[22]: True
Le code ci-dessus lèvera un TypeError si l'appel à sort_index est supprimé.
Plusieurs niveaux
Vous pouvez également empiler ou désempiler plusieurs niveaux à la fois en passant une liste de niveaux. Dans ce cas, le résultat final est le même que si chaque niveau de la liste était traité individuellement.
In [23]: columns = pd.MultiIndex.from_tuples([
....: ('A', 'cat', 'long'), ('B', 'cat', 'long'),
....: ('A', 'dog', 'short'), ('B', 'dog', 'short')],
....: names=['exp', 'animal', 'hair_length']
....: )
....:
In [24]: df = pd.DataFrame(np.random.randn(4, 4), columns=columns)
In [25]: df
Out[25]:
exp A B A B
animal cat cat dog dog
hair_length long long short short
0 1.075770 -0.109050 1.643563 -1.469388
1 0.357021 -0.674600 -1.776904 -0.968914
2 -1.294524 0.413738 0.276662 -0.472035
3 -0.013960 -0.362543 -0.006154 -0.923061
In [26]: df.stack(level=['animal', 'hair_length'])
Out[26]:
exp A B
animal hair_length
0 cat long 1.075770 -0.109050
dog short 1.643563 -1.469388
1 cat long 0.357021 -0.674600
dog short -1.776904 -0.968914
2 cat long -1.294524 0.413738
dog short 0.276662 -0.472035
3 cat long -0.013960 -0.362543
dog short -0.006154 -0.923061
La liste des niveaux peut contenir des noms de niveau ou des numéros de niveau (bien que les deux ne puissent pas être mélangés).
# df.stack(level=['animal', 'hair_length'])
#Ce code est égal à
In [27]: df.stack(level=[1, 2])
Out[27]:
exp A B
animal hair_length
0 cat long 1.075770 -0.109050
dog short 1.643563 -1.469388
1 cat long 0.357021 -0.674600
dog short -1.776904 -0.968914
2 cat long -1.294524 0.413738
dog short 0.276662 -0.472035
3 cat long -0.013960 -0.362543
dog short -0.006154 -0.923061
Données manquantes
Ces fonctions sont également flexibles dans la gestion des données manquantes et fonctionneront même si chaque sous-groupe de l'index hiérarchique n'a pas le même jeu d'étiquettes. Vous pouvez également gérer des index non triés (bien sûr, vous pouvez trier en appelant sort_index). Voici un exemple plus complexe:
In [28]: columns = pd.MultiIndex.from_tuples([('A', 'cat'), ('B', 'dog'),
....: ('B', 'cat'), ('A', 'dog')],
....: names=['exp', 'animal'])
....:
In [29]: index = pd.MultiIndex.from_product([('bar', 'baz', 'foo', 'qux'),
....: ('one', 'two')],
....: names=['first', 'second'])
....:
In [30]: df = pd.DataFrame(np.random.randn(8, 4), index=index, columns=columns)
In [31]: df2 = df.iloc[[0, 1, 2, 4, 5, 7]]
In [32]: df2
Out[32]:
exp A B A
animal cat dog cat dog
first second
bar one 0.895717 0.805244 -1.206412 2.565646
two 1.431256 1.340309 -1.170299 -0.226169
baz one 0.410835 0.813850 0.132003 -0.827317
foo one -1.413681 1.607920 1.024180 0.569605
two 0.875906 -2.211372 0.974466 -2.006747
qux two -1.226825 0.769804 -1.281247 -0.727707
Comme mentionné précédemment, vous pouvez appeler stack avec l'argument level pour choisir le niveau de la colonne à empiler.
In [33]: df2.stack('exp')
Out[33]:
animal cat dog
first second exp
bar one A 0.895717 2.565646
B -1.206412 0.805244
two A 1.431256 -0.226169
B -1.170299 1.340309
baz one A 0.410835 -0.827317
B 0.132003 0.813850
foo one A -1.413681 0.569605
B 1.024180 1.607920
two A 0.875906 -2.006747
B 0.974466 -2.211372
qux two A -1.226825 -0.727707
B -1.281247 0.769804
In [34]: df2.stack('animal')
Out[34]:
exp A B
first second animal
bar one cat 0.895717 -1.206412
dog 2.565646 0.805244
two cat 1.431256 -1.170299
dog -0.226169 1.340309
baz one cat 0.410835 0.132003
dog -0.827317 0.813850
foo one cat -1.413681 1.024180
dog 0.569605 1.607920
two cat 0.875906 0.974466
dog -2.006747 -2.211372
qux two cat -1.226825 -1.281247
dog -0.727707 0.769804
Si les sous-groupes n'ont pas le même jeu d'étiquettes, le dépilage peut entraîner des valeurs manquantes. Par défaut, les valeurs manquantes sont remplacées par les valeurs à remplir par défaut pour ce type de données (telles que «NaN» pour float, «NaT» pour datetime). Pour les types entiers, par défaut, les données sont converties en flottant et la valeur manquante est définie sur NaN.
In [35]: df3 = df.iloc[[0, 1, 4, 7], [1, 2]]
In [36]: df3
Out[36]:
exp B
animal dog cat
first second
bar one 0.805244 -1.206412
two 1.340309 -1.170299
foo one 1.607920 1.024180
qux two 0.769804 -1.281247
In [37]: df3.unstack()
Out[37]:
exp B
animal dog cat
second one two one two
first
bar 0.805244 1.340309 -1.206412 -1.170299
foo 1.607920 NaN 1.024180 NaN
qux NaN 0.769804 NaN -1.281247
ʻUnstack peut également prendre un argument optionnel fill_value` qui spécifie la valeur des données manquantes.
In [38]: df3.unstack(fill_value=-1e9)
Out[38]:
exp B
animal dog cat
second one two one two
first
bar 8.052440e-01 1.340309e+00 -1.206412e+00 -1.170299e+00
foo 1.607920e+00 -1.000000e+09 1.024180e+00 -1.000000e+09
qux -1.000000e+09 7.698036e-01 -1.000000e+09 -1.281247e+00
Pour MultiIndex
Même lorsque la colonne est «MultiIndex», elle peut être désempilée sans aucun problème.
In [39]: df[:3].unstack(0)
Out[39]:
exp A B ... A
animal cat dog ... cat dog
first bar baz bar ... baz bar baz
second ...
one 0.895717 0.410835 0.805244 ... 0.132003 2.565646 -0.827317
two 1.431256 NaN 1.340309 ... NaN -0.226169 NaN
[2 rows x 8 columns]
In [40]: df2.unstack(1)
Out[40]:
exp A B ... A
animal cat dog ... cat dog
second one two one ... two one two
first ...
bar 0.895717 1.431256 0.805244 ... -1.170299 2.565646 -0.226169
baz 0.410835 NaN 0.813850 ... NaN -0.827317 NaN
foo -1.413681 0.875906 1.607920 ... 0.974466 0.569605 -2.006747
qux NaN -1.226825 NaN ... -1.281247 NaN -0.727707
[4 rows x 8 columns]
Mise en forme par fusion
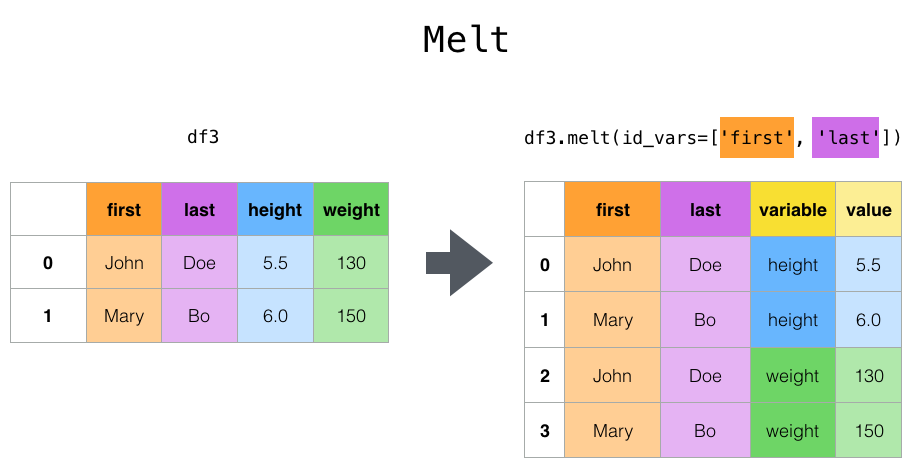
Fonction de niveau supérieur melt () et sa fonction DataFrame. melt () a un ou plusieurs DataFrame Convertit en un état "non pivoté" où il ne reste que deux colonnes non identifiantes, "variable" et "valeur", avec les colonnes * en tant que * variables d'identification * et toutes les autres colonnes en tant que * variables mesurées *. Utile pour. Les noms des colonnes restantes peuvent être personnalisés en spécifiant les paramètres var_name et value_name.
Par exemple
In [41]: cheese = pd.DataFrame({'first': ['John', 'Mary'],
....: 'last': ['Doe', 'Bo'],
....: 'height': [5.5, 6.0],
....: 'weight': [130, 150]})
....:
In [42]: cheese
Out[42]:
first last height weight
0 John Doe 5.5 130
1 Mary Bo 6.0 150
In [43]: cheese.melt(id_vars=['first', 'last'])
Out[43]:
first last variable value
0 John Doe height 5.5
1 Mary Bo height 6.0
2 John Doe weight 130.0
3 Mary Bo weight 150.0
In [44]: cheese.melt(id_vars=['first', 'last'], var_name='quantity')
Out[44]:
first last quantity value
0 John Doe height 5.5
1 Mary Bo height 6.0
2 John Doe weight 130.0
3 Mary Bo weight 150.0
Alternativement, wide_to_long () est un moyen pratique de convertir des données de panneau. C'est une fonction. Moins flexible que melt (), mais plus d'utilisateurs C'est sympa.
In [45]: dft = pd.DataFrame({"A1970": {0: "a", 1: "b", 2: "c"},
....: "A1980": {0: "d", 1: "e", 2: "f"},
....: "B1970": {0: 2.5, 1: 1.2, 2: .7},
....: "B1980": {0: 3.2, 1: 1.3, 2: .1},
....: "X": dict(zip(range(3), np.random.randn(3)))
....: })
....:
In [46]: dft["id"] = dft.index
In [47]: dft
Out[47]:
A1970 A1980 B1970 B1980 X id
0 a d 2.5 3.2 -0.121306 0
1 b e 1.2 1.3 -0.097883 1
2 c f 0.7 0.1 0.695775 2
In [48]: pd.wide_to_long(dft, ["A", "B"], i="id", j="year")
Out[48]:
X A B
id year
0 1970 -0.121306 a 2.5
1 1970 -0.097883 b 1.2
2 1970 0.695775 c 0.7
0 1980 -0.121306 d 3.2
1 1980 -0.097883 e 1.3
2 1980 0.695775 f 0.1
Combinaison avec statistiques et GroupBy
Vous devez être conscient que la combinaison de «pivot», «stack» et «unstack» avec GroupBy et les fonctions statistiques de base Series et DataFrame peut être une manipulation de données très expressive et rapide.
In [49]: df
Out[49]:
exp A B A
animal cat dog cat dog
first second
bar one 0.895717 0.805244 -1.206412 2.565646
two 1.431256 1.340309 -1.170299 -0.226169
baz one 0.410835 0.813850 0.132003 -0.827317
two -0.076467 -1.187678 1.130127 -1.436737
foo one -1.413681 1.607920 1.024180 0.569605
two 0.875906 -2.211372 0.974466 -2.006747
qux one -0.410001 -0.078638 0.545952 -1.219217
two -1.226825 0.769804 -1.281247 -0.727707
In [50]: df.stack().mean(1).unstack()
Out[50]:
animal cat dog
first second
bar one -0.155347 1.685445
two 0.130479 0.557070
baz one 0.271419 -0.006733
two 0.526830 -1.312207
foo one -0.194750 1.088763
two 0.925186 -2.109060
qux one 0.067976 -0.648927
two -1.254036 0.021048
#Des résultats similaires d'une autre manière
In [51]: df.groupby(level=1, axis=1).mean()
Out[51]:
animal cat dog
first second
bar one -0.155347 1.685445
two 0.130479 0.557070
baz one 0.271419 -0.006733
two 0.526830 -1.312207
foo one -0.194750 1.088763
two 0.925186 -2.109060
qux one 0.067976 -0.648927
two -1.254036 0.021048
In [52]: df.stack().groupby(level=1).mean()
Out[52]:
exp A B
second
one 0.071448 0.455513
two -0.424186 -0.204486
In [53]: df.mean().unstack(0)
Out[53]:
exp A B
animal
cat 0.060843 0.018596
dog -0.413580 0.232430
Tableau croisé dynamique
pivot () a différents types de données (chaînes de caractères) , Valeur numérique, etc.), mais pandas fournit pivot_table () pour pivoter en agrégeant des données numériques. api / pandas.pivot_table.html # pandas.pivot_table) est également fourni.
Tableau croisé dynamique de type feuille de calcul utilisant la fonction pivot_table () Peut être créé. Voir Cookbook pour des opérations plus avancées.
Cela prend quelques arguments.
--data: objet DataFrame.
--valeur: une colonne ou une liste de colonnes à agréger.
--ʻIndex: Colonne, Grouper, tableau de la même longueur que les données, ou une liste d'entre eux. Clés à regrouper par index dans le tableau croisé dynamique. Si un tableau est passé, il sera agrégé de la même manière que les valeurs de colonne. --columns: Colonnes, Grouper, tableaux de la même longueur que les données, ou une liste d'entre eux. Clés pour regrouper par colonnes dans le tableau croisé dynamique. Si un tableau est passé, il sera agrégé de la même manière que les valeurs de colonne. --ʻAggfunc: Fonction utilisée pour l'agrégation. La valeur par défaut est «numpy.mean».
Considérez l'ensemble de données suivant.
In [54]: import datetime
In [55]: df = pd.DataFrame({'A': ['one', 'one', 'two', 'three'] * 6,
....: 'B': ['A', 'B', 'C'] * 8,
....: 'C': ['foo', 'foo', 'foo', 'bar', 'bar', 'bar'] * 4,
....: 'D': np.random.randn(24),
....: 'E': np.random.randn(24),
....: 'F': [datetime.datetime(2013, i, 1) for i in range(1, 13)]
....: + [datetime.datetime(2013, i, 15) for i in range(1, 13)]})
....:
In [56]: df
Out[56]:
A B C D E F
0 one A foo 0.341734 -0.317441 2013-01-01
1 one B foo 0.959726 -1.236269 2013-02-01
2 two C foo -1.110336 0.896171 2013-03-01
3 three A bar -0.619976 -0.487602 2013-04-01
4 one B bar 0.149748 -0.082240 2013-05-01
.. ... .. ... ... ... ...
19 three B foo 0.690579 -2.213588 2013-08-15
20 one C foo 0.995761 1.063327 2013-09-15
21 one A bar 2.396780 1.266143 2013-10-15
22 two B bar 0.014871 0.299368 2013-11-15
23 three C bar 3.357427 -0.863838 2013-12-15
[24 rows x 6 columns]
Il est très facile de créer un tableau croisé dynamique à partir de ces données.
In [57]: pd.pivot_table(df, values='D', index=['A', 'B'], columns=['C'])
Out[57]:
C bar foo
A B
one A 1.120915 -0.514058
B -0.338421 0.002759
C -0.538846 0.699535
three A -1.181568 NaN
B NaN 0.433512
C 0.588783 NaN
two A NaN 1.000985
B 0.158248 NaN
C NaN 0.176180
In [58]: pd.pivot_table(df, values='D', index=['B'], columns=['A', 'C'], aggfunc=np.sum)
Out[58]:
A one three two
C bar foo bar foo bar foo
B
A 2.241830 -1.028115 -2.363137 NaN NaN 2.001971
B -0.676843 0.005518 NaN 0.867024 0.316495 NaN
C -1.077692 1.399070 1.177566 NaN NaN 0.352360
In [59]: pd.pivot_table(df, values=['D', 'E'], index=['B'], columns=['A', 'C'],
....: aggfunc=np.sum)
....:
Out[59]:
D ... E
A one three ... three two
C bar foo bar ... foo bar foo
B ...
A 2.241830 -1.028115 -2.363137 ... NaN NaN 0.128491
B -0.676843 0.005518 NaN ... -2.128743 -0.194294 NaN
C -1.077692 1.399070 1.177566 ... NaN NaN 0.872482
[3 rows x 12 columns]
L'objet résultat est un DataFrame qui a probablement un index hiérarchique sur les lignes et les colonnes. Si aucun nom de colonne n'est spécifié dans "values", le tableau croisé dynamique ajoutera un niveau de hiérarchie à la colonne pour contenir toutes les données pouvant être agrégées.
In [60]: pd.pivot_table(df, index=['A', 'B'], columns=['C'])
Out[60]:
D E
C bar foo bar foo
A B
one A 1.120915 -0.514058 1.393057 -0.021605
B -0.338421 0.002759 0.684140 -0.551692
C -0.538846 0.699535 -0.988442 0.747859
three A -1.181568 NaN 0.961289 NaN
B NaN 0.433512 NaN -1.064372
C 0.588783 NaN -0.131830 NaN
two A NaN 1.000985 NaN 0.064245
B 0.158248 NaN -0.097147 NaN
C NaN 0.176180 NaN 0.436241
Vous pouvez également utiliser «Grouper» pour les arguments «index» et «colonnes». Pour plus d'informations sur Grouper, voir Grouper avec Grouper (https://pandas.pydata.org/pandas-docs/stable/user_guide/groupby.html#groupby-specify).
In [61]: pd.pivot_table(df, values='D', index=pd.Grouper(freq='M', key='F'),
....: columns='C')
....:
Out[61]:
C bar foo
F
2013-01-31 NaN -0.514058
2013-02-28 NaN 0.002759
2013-03-31 NaN 0.176180
2013-04-30 -1.181568 NaN
2013-05-31 -0.338421 NaN
2013-06-30 -0.538846 NaN
2013-07-31 NaN 1.000985
2013-08-31 NaN 0.433512
2013-09-30 NaN 0.699535
2013-10-31 1.120915 NaN
2013-11-30 0.158248 NaN
2013-12-31 0.588783 NaN
Vous pouvez magnifiquement rendre la sortie d'une table avec des valeurs manquantes omises en appelant to_string si nécessaire.
In [62]: table = pd.pivot_table(df, index=['A', 'B'], columns=['C'])
In [63]: print(table.to_string(na_rep=''))
D E
C bar foo bar foo
A B
one A 1.120915 -0.514058 1.393057 -0.021605
B -0.338421 0.002759 0.684140 -0.551692
C -0.538846 0.699535 -0.988442 0.747859
three A -1.181568 0.961289
B 0.433512 -1.064372
C 0.588783 -0.131830
two A 1.000985 0.064245
B 0.158248 -0.097147
C 0.176180 0.436241
** pivot_table est également disponible comme méthode d'instance de DataFrame. ** **
⇒ DataFrame.pivot_table()。
Ajouter un sous-total
Passer margins = True à pivot_table ajoute une colonne et une ligne spéciales appelées ʻAll` qui agrège toute la catégorie de ligne et de colonne pour chaque groupe.
Tableau croisé
En utilisant crosstab (), vous pouvez en utiliser deux (ou plus) Vous pouvez croiser les éléments. Si aucun tableau de valeurs et de fonctions d'agrégation n'est passé, par défaut «tableau croisé» calculera le tableau de fréquence des éléments.
Il reçoit également les arguments suivants:
--ʻIndex: tableau etc. Valeurs à regrouper par ligne. --colonnes: tableau etc. Valeurs à regrouper par colonne. --valeurs: tableau etc. Optionnel. Un tableau de valeurs à agréger en fonction de l'élément. --ʻAggfunc: Fonction. Optionnel. S'il est omis, le tableau des fréquences est calculé.
--rownames: séquence. La valeur par défaut est «Aucun». Doit correspondre au nombre de tableaux de lignes passés (la longueur du tableau passé dans l'argument ʻindex). --colnames: séquence. La valeur par défaut est «Aucun». Doit correspondre au nombre de tableaux de colonnes passés (la longueur du tableau passé dans l'argument columns). --margins`: valeur booléenne. La valeur par défaut est «False». Ajoutez des sous-totaux aux lignes et aux colonnes.
- Valeur booléenne
normalize・ {"tous", "index", "colonnes"} ・ {0,1}. La valeur par défaut est «False». Normaliser en divisant toutes les valeurs par la somme des valeurs.
Sauf si un nom de ligne ou de colonne est spécifié dans le tableau croisé, l'attribut de nom de chaque «Série» transmise est utilisé.
Par exemple
In [65]: foo, bar, dull, shiny, one, two = 'foo', 'bar', 'dull', 'shiny', 'one', 'two'
In [66]: a = np.array([foo, foo, bar, bar, foo, foo], dtype=object)
In [67]: b = np.array([one, one, two, one, two, one], dtype=object)
In [68]: c = np.array([dull, dull, shiny, dull, dull, shiny], dtype=object)
In [69]: pd.crosstab(a, [b, c], rownames=['a'], colnames=['b', 'c'])
Out[69]:
b one two
c dull shiny dull shiny
a
bar 1 0 0 1
foo 2 1 1 0
Si crosstab ne reçoit que deux séries, une table de fréquences est renvoyée.
In [70]: df = pd.DataFrame({'A': [1, 2, 2, 2, 2], 'B': [3, 3, 4, 4, 4],
....: 'C': [1, 1, np.nan, 1, 1]})
....:
In [71]: df
Out[71]:
A B C
0 1 3 1.0
1 2 3 1.0
2 2 4 NaN
3 2 4 1.0
4 2 4 1.0
In [72]: pd.crosstab(df['A'], df['B'])
Out[72]:
B 3 4
A
1 1 0
2 1 3
Si les données transmises contiennent des données «catégoriques», les catégories ** toutes ** seront incluses dans le tableau croisé, même si les données réelles ne contiennent pas d'instances d'une catégorie particulière.
In [73]: foo = pd.Categorical(['a', 'b'], categories=['a', 'b', 'c'])
In [74]: bar = pd.Categorical(['d', 'e'], categories=['d', 'e', 'f'])
In [75]: pd.crosstab(foo, bar)
Out[75]:
col_0 d e
row_0
a 1 0
b 0 1
Normalisation
Le tableau des fréquences peut également être normalisé pour afficher des pourcentages au lieu des décomptes en utilisant l'argument «normaliser».
In [76]: pd.crosstab(df['A'], df['B'], normalize=True)
Out[76]:
B 3 4
A
1 0.2 0.0
2 0.2 0.6
«normaliser» peut également être normalisé pour chaque ligne ou colonne.
In [77]: pd.crosstab(df['A'], df['B'], normalize='columns')
Out[77]:
B 3 4
A
1 0.5 0.0
2 0.5 1.0
Si vous passez la troisième «Série» et la fonction d'agrégation («aggfunc») à «tableau croisé», la fonction pour la valeur de la troisième «Série» de chaque groupe défini dans les deux premières «Série». Est appliqué.
In [78]: pd.crosstab(df['A'], df['B'], values=df['C'], aggfunc=np.sum)
Out[78]:
B 3 4
A
1 1.0 NaN
2 1.0 2.0
Ajouter un sous-total
Enfin, vous pouvez ajouter des sous-totaux et normaliser leur sortie.
In [79]: pd.crosstab(df['A'], df['B'], values=df['C'], aggfunc=np.sum, normalize=True,
....: margins=True)
....:
Out[79]:
B 3 4 All
A
1 0.25 0.0 0.25
2 0.25 0.5 0.75
All 0.50 0.5 1.00
Binning
La fonction cut () effectue des calculs de regroupement pour les valeurs de tableau d'entrée. Je le ferai. Il est souvent utilisé pour convertir des variables continues en variables discrètes ou catégorielles.
In [80]: ages = np.array([10, 15, 13, 12, 23, 25, 28, 59, 60])
In [81]: pd.cut(ages, bins=3)
Out[81]:
[(9.95, 26.667], (9.95, 26.667], (9.95, 26.667], (9.95, 26.667], (9.95, 26.667], (9.95, 26.667], (26.667, 43.333], (43.333, 60.0], (43.333, 60.0]]
Categories (3, interval[float64]): [(9.95, 26.667] < (26.667, 43.333] < (43.333, 60.0]]
Passer un entier comme argument bins formera un bin de largeur égale. Vous pouvez également spécifier un bord de bac personnalisé.
In [82]: c = pd.cut(ages, bins=[0, 18, 35, 70])
In [83]: c
Out[83]:
[(0, 18], (0, 18], (0, 18], (0, 18], (18, 35], (18, 35], (18, 35], (35, 70], (35, 70]]
Categories (3, interval[int64]): [(0, 18] < (18, 35] < (35, 70]]
Si vous passez ʻIntervalIndex à l'argument bins`, il sera utilisé pour classer les données.
pd.cut([25, 20, 50], bins=c.categories)
Calcul des variables indicatrices et des variables fictives
Utiliser get_dummies () rendra les variables catégorielles "factices" et "marqueurs". Peut être converti en DataFrame. Par exemple, à partir d'une colonne «DataFrame» («Series») avec «k» différentes valeurs, vous pouvez créer un «DataFrame» contenant «k» colonnes constituées de 1 et de 0.
In [84]: df = pd.DataFrame({'key': list('bbacab'), 'data1': range(6)})
In [85]: pd.get_dummies(df['key'])
Out[85]:
a b c
0 0 1 0
1 0 1 0
2 1 0 0
3 0 0 1
4 1 0 0
5 0 1 0
Le préfixe des noms de colonne est utile, par exemple, lors de la fusion du résultat avec le DataFrame d'origine.
In [86]: dummies = pd.get_dummies(df['key'], prefix='key')
In [87]: dummies
Out[87]:
key_a key_b key_c
0 0 1 0
1 0 1 0
2 1 0 0
3 0 0 1
4 1 0 0
5 0 1 0
In [88]: df[['data1']].join(dummies)
Out[88]:
data1 key_a key_b key_c
0 0 0 1 0
1 1 0 1 0
2 2 1 0 0
3 3 0 0 1
4 4 1 0 0
5 5 0 1 0
Cette fonction est souvent utilisée avec des fonctions discrètes telles que «cut».
In [89]: values = np.random.randn(10)
In [90]: values
Out[90]:
array([ 0.4082, -1.0481, -0.0257, -0.9884, 0.0941, 1.2627, 1.29 ,
0.0824, -0.0558, 0.5366])
In [91]: bins = [0, 0.2, 0.4, 0.6, 0.8, 1]
In [92]: pd.get_dummies(pd.cut(values, bins))
Out[92]:
(0.0, 0.2] (0.2, 0.4] (0.4, 0.6] (0.6, 0.8] (0.8, 1.0]
0 0 0 1 0 0
1 0 0 0 0 0
2 0 0 0 0 0
3 0 0 0 0 0
4 1 0 0 0 0
5 0 0 0 0 0
6 0 0 0 0 0
7 1 0 0 0 0
8 0 0 0 0 0
9 0 0 1 0 0
Voir aussi Series.str.get_dummies S'il te plait donne moi.
get_dummies () peut également recevoir DataFrame. Par défaut, toutes les variables catégorielles (au sens statistique, c'est-à-dire celles avec des types de données * object * ou * categorical *) sont codées en variables factices.
In [93]: df = pd.DataFrame({'A': ['a', 'b', 'a'], 'B': ['c', 'c', 'b'],
....: 'C': [1, 2, 3]})
....:
In [94]: pd.get_dummies(df)
Out[94]:
C A_a A_b B_b B_c
0 1 1 0 0 1
1 2 0 1 0 1
2 3 1 0 1 0
Toutes les colonnes non-objet sont incluses dans la sortie telles quelles. Vous pouvez contrôler quelles colonnes sont encodées avec l'argument columns.
In [95]: pd.get_dummies(df, columns=['A'])
Out[95]:
B C A_a A_b
0 c 1 1 0
1 c 2 0 1
2 b 3 1 0
Vous pouvez voir que la colonne B est toujours incluse dans la sortie, mais elle n'est pas codée. Si vous ne voulez pas l'inclure dans la sortie, déposez B avant d'appeler get_dummies.
Comme pour la version Series, vous pouvez transmettre les valeurs de prefix et de prefix_sep. Par défaut, les noms de colonne sont utilisés comme préfixes et "_" est utilisé comme délimiteur de nom de colonne. prefix et prefix_sep peuvent être spécifiés des trois manières suivantes.
--String: utilisez la même valeur pour prefix ou prefix_sep dans chaque colonne à encoder.
--List: doit être aussi long que le nombre de colonnes à encoder.
--Dictionary: mappe les noms de colonnes en préfixes.
In [96]: simple = pd.get_dummies(df, prefix='new_prefix')
In [97]: simple
Out[97]:
C new_prefix_a new_prefix_b new_prefix_b new_prefix_c
0 1 1 0 0 1
1 2 0 1 0 1
2 3 1 0 1 0
In [98]: from_list = pd.get_dummies(df, prefix=['from_A', 'from_B'])
In [99]: from_list
Out[99]:
C from_A_a from_A_b from_B_b from_B_c
0 1 1 0 0 1
1 2 0 1 0 1
2 3 1 0 1 0
In [100]: from_dict = pd.get_dummies(df, prefix={'B': 'from_B', 'A': 'from_A'})
In [101]: from_dict
Out[101]:
C from_A_a from_A_b from_B_b from_B_c
0 1 1 0 0 1
1 2 0 1 0 1
2 3 1 0 1 0
Il peut être utile de ne conserver que le niveau k-1 de la variable catégorielle pour éviter la colinéarité lors de l'utilisation des résultats dans un modèle statistique. Vous pouvez passer à ce mode en utilisant drop_first.
In [102]: s = pd.Series(list('abcaa'))
In [103]: pd.get_dummies(s)
Out[103]:
a b c
0 1 0 0
1 0 1 0
2 0 0 1
3 1 0 0
4 1 0 0
In [104]: pd.get_dummies(s, drop_first=True)
Out[104]:
b c
0 0 0
1 1 0
2 0 1
3 0 0
4 0 0
Si la colonne ne contient qu'un seul niveau, elle sera omise dans le résultat.
In [105]: df = pd.DataFrame({'A': list('aaaaa'), 'B': list('ababc')})
In [106]: pd.get_dummies(df)
Out[106]:
A_a B_a B_b B_c
0 1 1 0 0
1 1 0 1 0
2 1 1 0 0
3 1 0 1 0
4 1 0 0 1
In [107]: pd.get_dummies(df, drop_first=True)
Out[107]:
B_b B_c
0 0 0
1 1 0
2 0 0
3 1 0
4 0 1
Par défaut, la nouvelle colonne sera «np.uint8» dtype. Utilisez l'argument dtype pour sélectionner un type de données différent.
In [108]: df = pd.DataFrame({'A': list('abc'), 'B': [1.1, 2.2, 3.3]})
In [109]: pd.get_dummies(df, dtype=bool).dtypes
Out[109]:
B float64
A_a bool
A_b bool
A_c bool
dtype: object
_ À partir de la version 0.23.0 _
Valeur d'élément (codage d'étiquette)
Pour encoder une valeur unidimensionnelle sous forme d'énumération, factorize ()Utilisez le.
In [110]: x = pd.Series(['A', 'A', np.nan, 'B', 3.14, np.inf])
In [111]: x
Out[111]:
0 A
1 A
2 NaN
3 B
4 3.14
5 inf
dtype: object
In [112]: labels, uniques = pd.factorize(x)
In [113]: labels
Out[113]: array([ 0, 0, -1, 1, 2, 3])
In [114]: uniques
Out[114]: Index(['A', 'B', 3.14, inf], dtype='object')
Notez que factorize est similaire à numpy.unique, mais gère NaN différemment.
: ballot_box_with_check: ** Remarque ** Le
numpy.uniquesuivant échoue avecTypeErrordans Python 3 en raison d'un bogue d'alignement. Pour plus d'informations, veuillez consulter ici.
In [1]: x = pd.Series(['A', 'A', np.nan, 'B', 3.14, np.inf])
In [2]: pd.factorize(x, sort=True)
Out[2]:
(array([ 2, 2, -1, 3, 0, 1]),
Index([3.14, inf, 'A', 'B'], dtype='object'))
In [3]: np.unique(x, return_inverse=True)[::-1]
Out[3]: (array([3, 3, 0, 4, 1, 2]), array([nan, 3.14, inf, 'A', 'B'], dtype=object))
: ballot_box_with_check: ** Remarque ** Si vous voulez traiter une colonne comme une variable catégorielle (comme le facteur R),
df [" cat_col "] = pd.Categorical (df [" col "]) ʻordf [" cat_col "] = Vous pouvez utiliser df ["col"] .astype ("category"). Pour une documentation complète sur [Categorical`](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.Categorical.html#pandas.Categorical), voir Introduction to Categorical //pandas.pydata.org/pandas-docs/stable/user_guide/categorical.html#categorical) et Documentation API Voir # api-arrays-categorical).
Exemple
Cette section contient des questions fréquemment posées et des exemples. Les noms de colonne et les valeurs de colonne associées ont des noms qui correspondent à la façon dont ce DataFrame est pivoté dans les réponses ci-dessous.
In [115]: np.random.seed([3, 1415])
In [116]: n = 20
In [117]: cols = np.array(['key', 'row', 'item', 'col'])
In [118]: df = cols + pd.DataFrame((np.random.randint(5, size=(n, 4))
.....: // [2, 1, 2, 1]).astype(str))
.....:
In [119]: df.columns = cols
In [120]: df = df.join(pd.DataFrame(np.random.rand(n, 2).round(2)).add_prefix('val'))
In [121]: df
Out[121]:
key row item col val0 val1
0 key0 row3 item1 col3 0.81 0.04
1 key1 row2 item1 col2 0.44 0.07
2 key1 row0 item1 col0 0.77 0.01
3 key0 row4 item0 col2 0.15 0.59
4 key1 row0 item2 col1 0.81 0.64
.. ... ... ... ... ... ...
15 key0 row3 item1 col1 0.31 0.23
16 key0 row0 item2 col3 0.86 0.01
17 key0 row4 item0 col3 0.64 0.21
18 key2 row2 item2 col0 0.13 0.45
19 key0 row2 item0 col4 0.37 0.70
[20 rows x 6 columns]
Pivoter avec un seul agrégat
Supposons que vous vouliez faire pivoter df de sorte que la valeur de col soit la colonne, la valeur de row soit l'index et la moyenne de val0 soit la valeur de la table. À ce stade, le DataFrame résultant ressemble à ceci:
col col0 col1 col2 col3 col4
row
row0 0.77 0.605 NaN 0.860 0.65
row2 0.13 NaN 0.395 0.500 0.25
row3 NaN 0.310 NaN 0.545 NaN
row4 NaN 0.100 0.395 0.760 0.24
Pour le trouver, utilisez pivot_table (). Notez que ʻaggfunc = 'mean'` est le comportement par défaut, bien que mentionné explicitement ici.
In [122]: df.pivot_table(
.....: values='val0', index='row', columns='col', aggfunc='mean')
.....:
Out[122]:
col col0 col1 col2 col3 col4
row
row0 0.77 0.605 NaN 0.860 0.65
row2 0.13 NaN 0.395 0.500 0.25
row3 NaN 0.310 NaN 0.545 NaN
row4 NaN 0.100 0.395 0.760 0.24
Vous pouvez également utiliser le paramètre fill_value pour remplacer les valeurs manquantes.
In [123]: df.pivot_table(
.....: values='val0', index='row', columns='col', aggfunc='mean', fill_value=0)
.....:
Out[123]:
col col0 col1 col2 col3 col4
row
row0 0.77 0.605 0.000 0.860 0.65
row2 0.13 0.000 0.395 0.500 0.25
row3 0.00 0.310 0.000 0.545 0.00
row4 0.00 0.100 0.395 0.760 0.24
Sachez également que vous pouvez transmettre d'autres fonctions d'agrégation. Par exemple, vous pouvez passer «sum».
In [124]: df.pivot_table(
.....: values='val0', index='row', columns='col', aggfunc='sum', fill_value=0)
.....:
Out[124]:
col col0 col1 col2 col3 col4
row
row0 0.77 1.21 0.00 0.86 0.65
row2 0.13 0.00 0.79 0.50 0.50
row3 0.00 0.31 0.00 1.09 0.00
row4 0.00 0.10 0.79 1.52 0.24
Une autre agrégation consiste à calculer la fréquence à laquelle les colonnes et les lignes apparaissent en même temps (appelée «agrégation croisée»). Pour ce faire, passez size au paramètre ʻaggfunc`.
In [125]: df.pivot_table(index='row', columns='col', fill_value=0, aggfunc='size')
Out[125]:
col col0 col1 col2 col3 col4
row
row0 1 2 0 1 1
row2 1 0 2 1 2
row3 0 1 0 2 0
row4 0 1 2 2 1
Pivoter avec plusieurs agrégats
Vous pouvez également effectuer plusieurs agrégations. Par exemple, vous pouvez passer une liste à l'argument ʻaggfunc` pour effectuer à la fois la somme totale et la moyenne de la moyenne.
In [126]: df.pivot_table(
.....: values='val0', index='row', columns='col', aggfunc=['mean', 'sum'])
.....:
Out[126]:
mean sum
col col0 col1 col2 col3 col4 col0 col1 col2 col3 col4
row
row0 0.77 0.605 NaN 0.860 0.65 0.77 1.21 NaN 0.86 0.65
row2 0.13 NaN 0.395 0.500 0.25 0.13 NaN 0.79 0.50 0.50
row3 NaN 0.310 NaN 0.545 NaN NaN 0.31 NaN 1.09 NaN
row4 NaN 0.100 0.395 0.760 0.24 NaN 0.10 0.79 1.52 0.24
Vous pouvez passer une liste au paramètre values pour l'agréger sur plusieurs colonnes de valeurs.
In [127]: df.pivot_table(
.....: values=['val0', 'val1'], index='row', columns='col', aggfunc=['mean'])
.....:
Out[127]:
mean
val0 val1
col col0 col1 col2 col3 col4 col0 col1 col2 col3 col4
row
row0 0.77 0.605 NaN 0.860 0.65 0.01 0.745 NaN 0.010 0.02
row2 0.13 NaN 0.395 0.500 0.25 0.45 NaN 0.34 0.440 0.79
row3 NaN 0.310 NaN 0.545 NaN NaN 0.230 NaN 0.075 NaN
row4 NaN 0.100 0.395 0.760 0.24 NaN 0.070 0.42 0.300 0.46
Vous pouvez transmettre une liste en tant que paramètre de colonne à subdiviser en plusieurs colonnes.
In [128]: df.pivot_table(
.....: values=['val0'], index='row', columns=['item', 'col'], aggfunc=['mean'])
.....:
Out[128]:
mean
val0
item item0 item1 item2
col col2 col3 col4 col0 col1 col2 col3 col4 col0 col1 col3 col4
row
row0 NaN NaN NaN 0.77 NaN NaN NaN NaN NaN 0.605 0.86 0.65
row2 0.35 NaN 0.37 NaN NaN 0.44 NaN NaN 0.13 NaN 0.50 0.13
row3 NaN NaN NaN NaN 0.31 NaN 0.81 NaN NaN NaN 0.28 NaN
row4 0.15 0.64 NaN NaN 0.10 0.64 0.88 0.24 NaN NaN NaN NaN
Développez les colonnes de la liste
_ À partir de la version 0.25.0 _
Les valeurs de colonne peuvent ressembler à une liste.
In [129]: keys = ['panda1', 'panda2', 'panda3']
In [130]: values = [['eats', 'shoots'], ['shoots', 'leaves'], ['eats', 'leaves']]
In [131]: df = pd.DataFrame({'keys': keys, 'values': values})
In [132]: df
Out[132]:
keys values
0 panda1 [eats, shoots]
1 panda2 [shoots, leaves]
2 panda3 [eats, leaves]
En utilisant [ʻexplode () ](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.Series.explode.html#pandas.Series.explode), values` Vous pouvez «exploser» les colonnes et convertir chaque élément de liste en une ligne distincte. Cela duplique la valeur d'index de la ligne d'origine.
In [133]: df['values'].explode()
Out[133]:
0 eats
0 shoots
1 shoots
1 leaves
2 eats
2 leaves
Name: values, dtype: object
Vous pouvez également développer les colonnes de DataFrame.
In [134]: df.explode('values')
Out[134]:
keys values
0 panda1 eats
0 panda1 shoots
1 panda2 shoots
1 panda2 leaves
2 panda3 eats
2 panda3 leaves
Series.explode () est une liste vide Remplacez par np.nan et conservez l'entrée scalaire. Le type de données Series résultant est toujours ʻobject`.
In [135]: s = pd.Series([[1, 2, 3], 'foo', [], ['a', 'b']])
In [136]: s
Out[136]:
0 [1, 2, 3]
1 foo
2 []
3 [a, b]
dtype: object
In [137]: s.explode()
Out[137]:
0 1
0 2
0 3
1 foo
2 NaN
3 a
3 b
dtype: object
À titre d'exemple typique, supposons qu'une colonne comporte une chaîne séparée par des virgules et que vous souhaitiez la développer.
In [138]: df = pd.DataFrame([{'var1': 'a,b,c', 'var2': 1},
.....: {'var1': 'd,e,f', 'var2': 2}])
.....:
In [139]: df
Out[139]:
var1 var2
0 a,b,c 1
1 d,e,f 2
Vous pouvez facilement créer un DataFrame vertical en effectuant des opérations d'expansion et de chaîne.
In [140]: df.assign(var1=df.var1.str.split(',')).explode('var1')
Out[140]:
var1 var2
0 a 1
0 b 1
0 c 1
1 d 2
1 e 2
1 f 2
Recommended Posts